分子生物学 第九章 DNA复制
1
2
3
4
5
6
7
8
9 ____ ___ __ ___ ____ __ __ ______
/\ _`\ /\_ \ __ /\ \__ __ /'___\ /\ _`\ /\ \/\ \/\ _ \
\ \ \L\ \ __ _____\//\ \ /\_\ ___ __ \ \ ,_\/\_\ ___ ___ ___ /\ \__/ \ \ \/\ \ \ `\\ \ \ \L\ \
\ \ , / /'__`\/\ '__`\\ \ \ \/\ \ /'___\ /'__`\ \ \ \/\/\ \ / __`\ /' _ `\ / __`\ \ ,__\ \ \ \ \ \ \ , ` \ \ __ \
\ \ \\ \ /\ __/\ \ \L\ \\_\ \_\ \ \/\ \__//\ \L\.\_\ \ \_\ \ \/\ \L\ \/\ \/\ \ /\ \L\ \ \ \_/ \ \ \_\ \ \ \`\ \ \ \/\ \
\ \_\ \_\ \____\\ \ ,__//\____\\ \_\ \____\ \__/.\_\\ \__\\ \_\ \____/\ \_\ \_\ \ \____/\ \_\ \ \____/\ \_\ \_\ \_\ \_\
\/_/\/ /\/____/ \ \ \/ \/____/ \/_/\/____/\/__/\/_/ \/__/ \/_/\/___/ \/_/\/_/ \/___/ \/_/ \/___/ \/_/\/_/\/_/\/_/
\ \_\
\/_/
DNA合成的化学基础
DNA合成需要脱氧核苷三磷酸和引物-模板接头
DNA合成必备的关键底物:
- 四种脱氧核糖核苷三磷酸: dNTP(dATP、dGTP、dCTP和dTTP)
- 引物-模板接头(primer-template junction): 具有特定排列的单链DNA或双链DNA。
两个重要部分
- 模板DNA: 单链DNA
- 引物: 与模板配对的短核苷酸链
DNA通过引物3’端的延伸进行合成
模板(template): 是指导所有互补脱氧核苷酸添加的单链DNA。
引物(primer): 是指一段较短的单链DNA或RNA, 它能与DNA的一条链配对提供3’-OH末端以作为DNA聚合酶合成脱氧核苷酸链的起始点。
合成方向: 5’->3’
焦磷酸水解是DNA合成的驱动力
DNA合成的驱动力:
引物链3’端的羟基基团攻击引入的核苷三磷酸α-磷酰基基团, 导致引入的引物3’端延伸一个核苷酸并释放一个焦磷酸分子。叫磷酸酯酶将焦磷酸水解成2个磷酸基团为DNA合成提供能量。
DNA聚合酶的作用机制
DNA聚合酶用一个活性位点催化DNA的合成
DNA聚合酶的检测机制:
- DNA聚合酶催化核苷酸添加需要正确配对的碱基: 只有形成正确的碱基对, 引物3’-OH和在最佳催化位置上的引入核苷三磷酸的α-磷酸才能发生催化反应。
- 空间位阻阻止DNA聚合酶催化反应使用rNTP: DNA聚合酶中核苷酸结合口袋非常小, 不能容纳rNTP上的2’-OH。
DNA聚合酶像手一样握住引物-模板接头
DNA聚合酶的结构域:
DNA聚合酶的3个结构域被称为拇指、手指和手掌。
手掌域
手掌域由一个β折叠片构成, 含有催化位点的基本元件, 尤其是DNA聚合酶的这一区域结合2个二价金属离子(镁离子和锌离子), 可以改变正确碱基配对的dNTP和引物3’-OH周围的化学环境。除了催化作用外, 手掌域还负责检查最新加入的核苷酸碱基配对的准确性。
作用机制
- 聚合酶活性位点: 第一个金属离子降低3’-OH对氢的亲和力, 产生一个准备引入dNTP上α-磷酸进行亲核攻击的3’O-;第二个金属离子与dNTP的β-和γ-磷酸负电荷协同作用, 稳定由引物和引入核苷酸连接在一起时产生的焦磷酸。
- 核酸外切酶活性位点: 与新合成DNA小沟中的正确配对的碱基对形成大量的氢键, 这一接触无碱基特异性。错误配对的DNA影响小沟结合形, 使催化活性显著降低, 同时DNA聚合酶对新合成DNA降低亲和力使引物-模板从聚合酶活性位点脱离转向一个独立的、具有校对功能的核酸酶活性位点上将错配的DNA切除。
功能
- 有两个催化位点, 一个催化添加新的dNTP合成位点, 另一个是把错配的dNTP去除, 起校对功能;
- 聚合位点有两个金属离子, 它们有利于催化的进行;
- 检测配对的准确性。
手指域
手指域中的几个残基可与引入的dNTP结合。一旦引入的dNTP与模板之间形成正确的碱基配对, 手指域即发生移动, 包围住dNTP。
功能
- 使引入的核苷酸与催化性的金属离子密切接触, 促进催化反应;
- 使模板的磷酸二酯键骨架在第一个和第二个碱基间产生约90°回折, 使催化位点上引物后即将配对的模板碱基暴露, 避免了下一个核苷酸添加时可能会造成配对的模板碱基选择混淆。
拇指域
拇指域与催化的关联不紧密, 而是与最新近合成的DNA相互作用。
功能
- 不直接参与催化, 但可维持引物以及活性部位的正确位置;
- 帮助维持DNA聚合酶与其底物之间的紧密连接。
DNA聚合酶是一种延伸酶
延伸能力(processivity): 是酶处理多聚底物时的一种特性。对于DNA聚合酶来说, 延伸能力定义为每次酶与引物-模板接头结合时所添加核苷酸的平均数, 每个DNA聚合酶都有其特征性的延伸能力。DNA合成的速度主要取决于DNA聚合酶的延伸能力。
DNA聚合酶的催化是快速的。最快的DNA聚合酶能在引物链上每秒添加1000个核苷酸。
外切核酸酶校正新合成出的DNA
影响DNA聚合酶精确度的主要因素是: 碱基偶然变换成互变异构体。
校正外切核酸酶(proofreading exonuclease): 这类酶可以从DNA的3’端, 即从新DNA链的生长端开始降解DNA。
内切核酸酶(endonuclease): 在DNA链中部切割的核酸酶。
校正外切核酸酶的作用机制: 在DNA合成时, 无需DNA脱离聚合酶即可进行校正。碱基错配的引物附近添加核苷酸的能力降低, 增加了校正外切核酸酶的活性。不正确的核苷酸被外切酶移除, 正确配对的引物-模板接头滑回DNA聚合酶的活性位点, DNA合成继续进行。
三个水平保证DNA合成的准确性:
- DNA聚合酶的监控机制使得每添加$10^5$个核苷酸插入一个不正确的核苷酸;
- 校正外切核酸酶将错配碱基的发生概率降低到每添加$10^7$个核苷酸出现一次错误;
- 复制后错配修复过程将发生概率降低到$10^{10}$个核苷酸出现一次错误。
复制叉
复制叉上DNA的两条链同时合成
复制叉(replication fork): 复制时, 双链DNA要解开成两股链分别进行DNA合成, 刚分开的模板链与未复制的双链DNA之间的连接区呈叉子形状, 被称为复制叉。
前导链(leading strand): 在DNA复制过程中, 与复制叉运动方向相同, 以5’->3’方向连续合成的链被称为前导链。当DNA模板链一解开就可以开始合成前导链。
后随链(lagging strand): 在DNA复制过程中, 与复制叉运动方向相反, 以3’->5’方向以不连续的方式延伸的DNA链被称为后随链或滞后链, 等复制叉运动并暴露出足够长的模板后才能进行合成。
冈崎片段(Okazaki fragment): 在后随链上形成的新链DNA短片段称为冈崎片段, 在不同生物中冈崎片段长度有所不同。前后两个冈崎片段通过DNA连接酶共价连接形成一条完成的新DNA链。冈崎片段是DNA复制中短暂的中间产物。
DNA新链的起始需要一条RNA引物
为什么DNA聚合酶需要带有游离3’-OH的引物: DNA聚合酶不能从头起始新DNA链的合成, RNA聚合酶可以从头起始新RNA链的合成。
引物酶(primase): 是一种能在单链DNA模板上制造短RNA引物(5~10个核苷酸长度)的特殊的RNA聚合酶。不需要用特异DNA序列来起始新RNA引物的合成。引物酶与DNA解旋酶结合时引物酶的活性会显著提高。前导链和后随链都需要引物酶来起始DNA合成
完成DNA复制必须除去RNA引物
RNA引物的去除:
- RNA酶H(RNase H): 能够识别并除去各条RNA引物的大部分。RNase H只能断裂两个核糖核苷酸之间的键, 因此除了与DNA末端直接连的核糖核苷酸之外, 此酶能特异性降解与DNA碱基配对的RNA。
- 最后一个核糖核苷酸是由从5’外切核酸酶除去的。
- 在原核生物中, 后随链上的引物由DNA PolI切除并补齐。
DNA连接酶(DNA ligase): 是生物体内重要的酶, 其所催化的反应在DNA的复制和修复过程中起着重要的作用。连接DNA链3’-OH末端和, 另一DNA链的5’-P末端, 使二者生成磷酸二酯键, 从而把两段相邻的DNA链连成完整的链。连接酶的催化作用需要消耗ATP。
DNA解旋酶在复制叉之前解开双螺旋
DNA解旋酶(DNA helicase): 在复制叉上, 催化双链DNA两条链的分离的一组酶称为DNA解旋酶。解旋酶沿单链DNA的运动需要的能量由ATP来提供。每个DNA解旋酶在单链DNA上都沿着一定的方向运动。所有DNA解旋酶都具有极性。DNA解旋酶可以有5’→3’或3’→5’的活性, 方向根据结合的那条链决定。
复制前单链结合蛋白使单链DNA稳定
单链DNA结合蛋白(ssDNA-bindingprotein, SSB): 一种与单链DNA紧密结合的蛋白, 它的结构可以防止复制叉处单链DNA本身重新形成双链。它只起到维持单链的作用, 并不能起解链的作用。一个SSB的结合会促进另一个SSB与其紧邻的单链DNA结合。
协同结合(cooperative binding): 一个单链DNA结合蛋白结合单链DNA会促进另一个单链DNA结合蛋白结合上来, 这一过程成为协同结合。SSB分子之间相互结合大大稳定了SSB与单链DNA之间的相互作用, 使得已有一个或多个SSB结合位点比其他位点更易结合新的SSB。
拓扑异构酶除去DNA解旋时在复制叉上产生的超螺旋
复制过程中存在的问题:
随着复制叉上DNA链的分离, 叉前的双链DNA变得更加正超螺旋化。如果DNA链保持不断裂, 则不会减少DNA双链解旋的连环数(两条DNA互绕的次数)。随着DNA解旋酶的行进, DNA必须在越来越少的碱基对中维持同样数目的连环数。如果没有机制来消除这些累积的超螺旋, 复制机器将在不断上升的压力面前陷于停顿。
对于细菌的环形染色体, 此问题非常明显, 但真核生物染色体也有同样的问题。
由DNA解旋酶的作用所引入的超螺旋, 可通过拓扑异构酶(topoisomerase)对在复制叉前未被复制的双链DNA的作用来消除。这些酶是通过断裂一条或两条DNA链但不让其脱落, 并且让同等数目的DNA链通过断口来完成的。通过这种方式, 快速地消除了DNA解旋所产生的超螺旋累积。
复制叉酶扩大了DNA聚合酶底物的范围
DNA聚合酶本身只能使与单链DNA模板退火的3’-OH引物延长。引物酶、DNA解旋酶及拓扑异构酶的加入极大地扩大了DNA聚合酶可能的底物范围。
为什么说复制叉酶扩大了DNA聚合酶底物范围?
- 引物酶提供了在任何单链DNA上都能起始新的DNA链的能力。引物酶也要求除去RNA引物来完成复制。
- DNA解旋酶对链的解离, 以及拓扑异构酶对正超螺旋的消除使DNA聚合酶能够复制双链DNA。
- 在复制叉上工作的蛋白质与DNA密切作用, 但采用的是序列非特异的方式。这些相互作用利用DNA与特定碱基对无关的特性:负电荷及磷酸骨架结构(如DNA聚合酶的拇指域)、小沟内残基的氢键(如DNA聚合酶的手掌域)、碱基之间的疏水堆积作用(如SSB)。
- 很多蛋白质具有环绕(如DNA解旋酶)或包裹(如DNA聚合酶)DNA的结构, 以保持与DNA的结合。
DNA聚合酶的特化
细胞中DNA聚合酶为形式不同的功能而被特化
E. coli中至少有5种DNA聚合酶。
DNA聚合酶III全酶(DNA Pol III holoenzyme): 负责E.coli基因组复制的蛋白质复合物, 有校正外切酶的活性。具有高度延伸能力。DNA Pol III全酶含有3个拷贝的核心DNAPoI III和1个拷贝的五亚基滑动夹装载器。虽然在全酶中滑动夹装载器只有一个拷贝, 但包括3个拷贝τ蛋白, 每个τ蛋白结合一个DNAPol III核心酶。
DNA聚合酶I(DNAPol I): 专门用于除去起始DNA合成所需的RNA引物。DNA聚合酶I具有5’外切核酸酶的活性, 能将DNA合成位点最上游的RNA或DNA迅速除去。DNA聚合酶I的延伸能力不强, 每次结合仅能添加20~ 100个核苷酸, 使其适用于RNA引物(可以除去RNase H所不能除去的RNA-DNA连接)的去除和在所产生的单链DNA缺口中进行DNA的合成。
真核细胞中对与基因组的复制至关重要的有3种: DNA Polδ、DNA Polε及DNA Polα/引物酶。这些真核生物的DNA聚合酶由多个亚基构成。
聚合酶切换(polymerase switching): DNA Polα/引物酶参与新DNA链的起始, 引物酶合成RNA引物后, 所产生的RNA引物-模板接头立即与DNAPolα结合以启动DNA的合成, 由于DNAPolα/引物酶的延伸能力相对较低, 很快就被高延伸性的DNAPolδ或ε取代。这一过程称为聚合酶的切换。
在真核细胞复制叉上有3种不同的DNA聚合酶在工作。DNAPolδ和ε在复制叉上特异性地复制不同的链, DNA Polδ合成前导链, DNA Polε合成后随链。
滑动夹极大地增加了DNA聚合酶的延伸能力
为什么DNA聚合酶具有较高的延伸性?
- DNA聚合酶在复制叉上具有高延伸能力的关键原因在于它们与被称为滑动DNA夹的蛋白质间的结合。在无滑动夹的情况下, DNA聚合酶平均每合成20~100个碱基对就从DNA模板上解离并散落开来。在有滑动夹的情况下, DNA聚合酶的活性部位仍经常从DNA的3’-OH末端脱落, 但是其与滑动夹的结合阻止了聚合酶从DNA上散开。通过保持DNA聚合酶与DNA的紧密接触, 滑动夹使DNA聚合酶能迅速地重新结合到同一引物模板接头上, 大大增加了DNA聚合酶的延伸能力。
- DNA聚合酶拇指域与最新合成的DNA相互作用, 维持引物和活性位点的正确位置, 帮助维持DNA聚合酶与其底物之间的较强的结合, 使DNA聚合酶每次与引物-模板接头结合时, 具有添加多个dNTP的能力, 因此提高了DNA聚合酶的延伸能力。
- DNA聚合酶沿DNA滑行的能力有利于提高延伸能力, 其中有两种利于DNA聚合酶沿DNA滑行的相互作用: 拇指域和磷酸骨架间静电作用(序列非特异性)、手掌域与小沟间的氢键作用(序列非特异性, 每添加一个核苷酸断裂一次)。
滑动DNA夹(sliding DNA clamp): 滑动夹蛋白是病毒、细菌、酵母及人类等生物DNA复制体系中的保守部件, 夹子均为同样的六边形对称并有相同的直径但亚基数目却不相同, 这些亚基组装成”油炸圈饼”的形状。DNA聚合酶与滑动夹所形成的复合体在DNA合成时沿着DNA模板高效地移动, 提高DNA聚合酶的延伸性。
DNA聚合酶如何摆脱滑动夹?
当单链DNA模板完全复制后, DNA聚合酶必须从双链DNA和滑动夹上释放, 与新的引物模板接头结合。此释放是通过DNA聚合酶与滑动夹之间亲和力的变化实现的。与引物模板接头结合的DNA聚合酶与夹子有很高的亲和力。当DNA聚合酶到达单链DNA模板的末端时(如冈崎片段的末端), 活性部位存在的双链DNA使DNA聚合酶构象发生变化, 降低了其与滑动夹及DNA之间的亲和力。当聚合酶完成一段DNA复制后, 它即从滑动夹释放, 在新的引物-模板接头上开始工作。
释放DNA聚合酶后, 滑动夹不立即从复制的DNA上脱落下来。相反, 其他必须在DNA新合成处作用的蛋白质通过与夹子蛋白相互作用而执行其功能。真核细胞中组装染色质的酶通过与真核滑动DNA夹(称为PCNA)滑动DNA夹(sliding DNA clamp): 滑动夹蛋白是病毒、细菌、酵母及人类等生物DNA复制体系中的保守部件, 夹子均为同样的六边形对称并有相同的直径但亚基数目却不相同, 这些亚基组装成”油炸圈饼”的形状。DNA聚合酶与滑动夹所形成的复合体在DNA合成时沿着DNA模板高效地移动, 提高DNA聚合酶的延伸性。
滑动夹通过滑动夹装载器打开并安置在DNA上
滑动夹装载器(sliding clamp loader): 一类特殊的蛋白复合体催化滑动夹的打开并将其安放在DNA上。这些酶通过结合ATP并将其水解而将滑动夹环置于DNA的引物-模板接头上。滑动夹装载器还能将滑动夹在不使用的时候将其从DNA上移除, 这一过程不需要ATP。这些酶改变其靶物(滑动夹)的构象, 但并不改变其化学组成。
控制滑动夹从DNA上装载和移除过程:
- 装载: 只要细胞中出现引物模板接头,滑动夹的装载就可能发生。此DNA结构并非只在DNA复制时形成, 在DNA修复事件中也有。
- 移除: 滑动夹只有当其不再被其他酶使用时才能从DNA上移除。滑动夹装载器和DNA聚合酶不能同时与滑动夹作用, 因为它们在滑动夹的同一面上有重叠的结合位点。因此, 与DNA聚合酶结合的滑动夹不会从DNA上移除。核小体组装因子、冈崎片段修复蛋白及其他DNA修复蛋白均与夹子装载器一样, 与滑动夹的同一区域作用, 因此, 滑动夹只能在与其相作用的所有酶都完成工作后才能从DNA上移除。
复制叉上的DNA合成
在复制叉上前导链和后随链同时进行合成, 这对在DNA合成期内限制细胞内单链DNA的数量有重要意义。当DNA的单链区断裂时, 在染色体上会产生完全的断裂, 这比双链DNA上的一条单链DNA断裂修复起来要困难得多。而且, 修复这种类型的损伤经常会导致DNA的突变。限制DNA处于此状态的时间是十分重要的。为了协调DNA两条链的复制, 在复制叉上有多种DNA聚合酶在行使功能。
长号模型
“长号模型”: 本模型描述了两个后随链DNA聚合酶交替启动冈崎片段的合成。
- E. coli DNA复制叉上的DNA解旋酶在后随链模板上沿着5’→3方向运动。DNA PolIII全酶通过τ亚基与DNA解旋酶相互作用, τ亚基也与DNA Pol II I核心酶结合。一个DNA Pol III核心酶复制前导链, 另两个DNA Pol III核心酶复制后随链。SSB覆盖在DNA的单链区域。
- DNA引物酶周期性地与DNA解旋酶结合并在后随链模板上合成新的RNA引物。
- 新的RNA引物合成后, DNA滑动夹装载器就在模板引物接头处加载滑动夹。
- 空闲的”第二个”后随链DNA聚合酶在模板-引物接头处快速识别DNA滑动夹, 并合成新的冈崎片段。
- 当”第一个”后随链DNA聚合酶到达冈崎链模板的末端后, 从滑动夹释放出来。解离的DNAPolII核心酶通过滑动夹装载器的τ亚基与解旋酶保持着联系, 所以在加载滑动夹后该DNA聚合酶立即与RNA引物-模板接头在理想的位置结合。
复制叉蛋白之间的相互作用形成E.coli的复制体
复制叉上的蛋白质之间的相互作用: 除了DNA Pol III全酶组分之间的相互作用以外, 还有几个复制叉蛋白-蛋白间的相互作用加快了复制叉的进程。
其中最重要的是DNA解旋酶与DNAPoL III全酶之间的相互作用。该作用由组成全酶的滑动夹装载器的τ亚基介导, 将解旋酶和DNA Pol III全酶连接到一起。此作用使解旋酶运动的速度增加10倍, 从而激发了解旋酶的活性。如果DNA解旋酶与DNA聚合酶分离, 其速度将减慢。解旋酶活性与DNAPolI耦联, 防止了解旋酶从DNAPol I全酶上”逃脱”。
第二个重要的蛋白质间相互作用发生在DNA解旋酶与引物酶之间。引物酶与复制叉的结合并不紧密。在约1秒一次的间隔中, 引物酶与解旋酶及SSB覆盖的单链DNA结合并合成新的RNA引物。虽然DNA解旋酶与引物酶之间的作用相对较弱, 但是其相互作用大大激发了引物酶的活性(提高约1000倍)。RNA引物合成后, 引物酶从DNA解旋酶上脱落并进入溶液中。在E.coli中, 引物酶与DNA解旋酶之间相对较弱的相互作用对于调节冈崎片段长度很重要。紧密的结合可导致后随链上的引物合成更频繁, 这样冈崎片段就会较短。较弱的作用则产生较长的冈崎片段。
复制体(replisome): 复制叉上发挥作用的全部蛋白质的总和称为复制体。包含DNA聚合酶、解旋酶、单链结合蛋白和其他辅助因子。这些蛋白质在一起形成调度有序的”工厂”, 从而进行DNA复制的聚合反应。
DNA复制的起始
特定的基因组DNA序列指导DNA复制的起始
复制起始位点(origin of replication): 在DNA上发生DNA解旋及DNA合成起始特殊位点称作复制起始位点。其数目依不同生物而不同, 每个染色体上可有少至一个、多至数千个起始位点。
复制起始的复制子模型
复制子(replicon): 单独复制的一个DNA单元被称为一个复制子。一个复制中在任何一个细胞周期内只能复制一次。E.coli整个染色体是一个复制子;每个真核染色体存在多个复制子。复制子模型包括控制复制起始的两个元件: 复制器和起始子。
复制器(replicator): 是指足够指导DNA复制起始的整套顺式作用的DNA序列。复制器的DNA序列的两个共同特性: ①它们含有起始子蛋白结合位点, 此位点是组装复制起始机器的核心;②它们含有一段富含AT的DNA,此段DNA容易解旋但并不自发进行。
起始子蛋白(initiator): 复制子模型中的元件之一, 此蛋白质特异性地识别复制器中的一个DNA元件并激活复制子中的的起始。在起始子蛋白是细菌复制起始中唯一涉及的序列特异性DNA结合蛋白。而大部分真核细胞, 起始子蛋白没有表现出序列特异性结合活性。
E.coli起始子蛋白是DnaA, 真核细胞中, 起始子是一个被称为ORC的六蛋白复合体。起始子蛋白功能:
- 这些蛋白质与复制器中的特异DNA序列结合;
- 一旦与DNA结合, 它们就扭曲或解旋其结合位点附近的DNA区域;
- 起始子蛋白与复制起始所需的其他因子相互作用, 使它们聚集到复制器上。
复制器序列包括起始子结合位点和容易解旋的DNA
大肠杆菌复制器结构: E.coli染色体复制所需要的单个复制器被称为”oriC”。有两个重复的基序对oriC功能至关重要。9mer单位的基序是E.coli起始子DnaA的结合位点, 其在oriC上重复5次。13mer单位的基序重复3次, 是起始时单链DNA形成的起始位点。”
结合和解旋: 起始子蛋白对复制起始位点的选择激活
蛋白质-蛋白质和蛋白质-DNA相互作用指导起始过程
DNA复制的起始过程:
E. coli起始子蛋白DnaA与oriC中重复的9核苷酸单位元件结合并受ATP的调控。DnaA还会在复制器所形成的单链DNA上聚集其他的包括DNA解旋酶的复制蛋白。
真核细胞中, 起始子是一个被称为起始位点识别复合体(origin recognition complex, ORC)的六蛋白复合体。ORC可识别酵母复制器上称为A元件的保守序列, 以及次保守的B1元件。ORC可将其他复制蛋白全部召集到复制器上。ORC具有起始子三项常见功能中的两项: 与复制子结合并将其他复制蛋白召集到复制器上。
E.coli复制起始过程:
- 多个DnaA-ATP蛋白结合到oriC内的9mer重复序列。DnaA-ATP与这些序列的结合导致链在13mer重复处分离。
- DNA解旋酶(DnaB)和DNA解旋酶装载器(DnaC)形成复合物与DnaA起始位点结合。DNA解旋酶装载器催化DNA解旋酶蛋白环的打开和环在单链DNA起始位点位置上的套装。
- 每个DNA解旋酶募集一个引物酶, 引物酶在各模板上合成RNA引物。RNA引物导致解旋酶装载器从解旋酶上释放, 激活解旋酶。DNA解旋酶的运动将所有剩下的、与复制器结合的DnaA除去。
- 新合成的引物和解旋酶被DNAPol III全酶中的夹子装载器元件识别。滑动夹在每条RNA引物上组装, 前导链的合成由全酶的三个核心DNA Pol III中的一个启动。
- 当各个DNA解旋酶运动约1000bp之后, 在各自的后随链模板上合成第二条RNA引物并装载滑动夹。产生的引物-模板接头被全酶中的第二个DNA Pol III核心酶识别, 导致后随链合成的启动。
- DNA引物酶周期性地与DNA解旋酶结合并在后随链模板上合成新的RNA引物。新的RNA引物合成后, DNA滑动夹装载器就在模板引物接头处加载滑动夹。第三个DNA Pol III核心酶在模板-引物接头处快速识别DNA滑动夹, 并合成新的冈崎片段。
真核染色体每一个细胞周期精确地复制一次
真核染色体复制每个细胞周期复制一次的前提条件:
- 必须激活足够多的起始位点, 以保证每个S期中每条染色体都被完全复制。通常, 完成复制并不需要把所有的起始位点都激活, 但是如果激活得太少, 基因组中将有部分区域没有被复制。
- 要保证任何一个已复制的复制起始位点不可重启动。一个起始位点不管是被激活而使其自身复制, 还是被邻近起始位点产生的复制又所复制, 它都必须失活, 直至下一轮细胞分裂开始为止。
装载解旋酶是真核细胞中复制起始的第一步
真核细胞中复制起始需要的两个步骤:
- 复制器的选择
- 起始位点的激活。发生在细胞周期不同时期。
复制起始步骤:
- 1.复制器的选择—真核细胞装载解旋酶(发生于G1期):
- 1.真核生物DNA复制解旋酶的装载是一个有序的过程, 由ATP结合的起始位点识别复合体(ORC)与复制器的结合引发。
- 2.ORC与复制器结合后, 就募集ATP结合的Cdc6和两个拷贝的Mcm2-7解旋酶并结合到解旋酶装载蛋白Cdtl上。
- 3.这些蛋白的组装触发Cdc6水解ATP,导致了Mcm2-7头对头二聚体的装载, 以致它们将起始点DNA双链起点包围起来, 并使得Cdc6和Cdtl从起始点上被释放。
- 4.ORC水解ATP可以重置该过程。
- 5.ATP和ADP的交换允许新一轮解旋酶的加载。
- 2.起始位点的激活——真核复制体组装(发生在S期):
- 1.当细胞进入细胞周期的S期时, 两种蛋白激酶——CDK和DDK被激活。
- 2.DDK使装载的Mcm2-7解旋酶磷酸化, 而CDK使Sld2和Sld3磷酸化。
- 3.磷酸化的Sld2和Sld3与Dpb11结合, 这些蛋白质导致解旋酶激活蛋白Cdc45和GINS与解旋酶结合。Cdc45和GINS与Mcm2-7解旋酶形成一种稳定的复合物(称为CMG复合体), 并极大地促进了Mcm2-7解旋酶的活性。DNA解旋之前, 前导链DNA聚合酶(ε)被招募到此时的解旋酶上。
- 4.在CMG复合物形成后, SId2、SId3和Dpb11从起始位点释放。DNA Polα/引物酶和DNAPolδ(主要作用于后随链)只能在DNA解旋后被召募。
对解旋酶装载和激活的调控使每个细胞周期中仅有一轮复制发生
为什么真核染色体每一个细胞周期精确地复制一次?
在每个细胞周期中发生的两种复制状态之间的相互转换。
在G1期,细胞处于解旋酶装载阶段,可以进行解旋酶的装载但是不能激活已组装好的解旋酶;进入S期以及之后的G2和M期时,在G1期装载的解旋酶可以被激活,而新的解旋酶的装载则受到了严格的抑制。这是受到CDK激酶的功能调控的。
这些酶在调控复制时的功能是相互对立的:
- 这些酶是激活装载的解旋酶从而起始DNA的复制;
- CDK的活性抑制了解旋酶的装载。
在G1期时CDK的水平很低, 因而可以允许解旋酶的装载, 但是阻止解旋酶的激活。
进入S期伴随着CDK活性的迅速升高, 从而激活装载好的解旋酶同时阻止新的解旋酶的装载。
在细胞周期的剩余时期(S期、G2期和M期),CDK的水平依然是升高的, 高活性水平的CDK阻止了ORC、Cdc6和Cdtl的功能, 从而抑制这些时期解旋酶的装载, 没有新的解旋酶复合体形成, 复制重新起始是不可能发生的, 从而保证了染色体每个细胞周期仅复制一次。
只有当细胞将其染色体分离且完成细胞分裂的时候, CDK的活性才消失, 新一轮的解旋酶装载才能开始。
真核和原核生物DNA复制起始的相似性
共同之处:
- 第一步是起始子蛋白对复制器的识别。起始子蛋白和一个或多个解旋酶装载蛋白共同将 DNA 解旋酶募集到复制器上。
- 解旋酶产生单链 DNA 区域,作为 RNA 引物合成的模板。
- 引物合成后,复制体的其他元件通过与形成的引物-模板接头相互作用进行组装。
不同之处:
- 与真核细胞不同,快速分裂的细菌细胞在一次细胞循环内多次起始复制
- 复制调节中最严谨的步骤在真核细胞和原核细胞中也是不同的。真核细胞对复制的调节集中在MCM解旋酶装载到 DNA 的起始位点上,而细菌细胞集中在 DnaA 起始蛋白与 DNA 的结合上。
结束复制与末端复制问题
子代DNA分子的分离需要拓扑异构酶II
如何解决环形染色体复制结束问题?
环形染色体复制完成之后, 产生的子代DNA分子如铰链般连接在一起。为了使这些染色体分离到子细胞中去, 两个环形DNA分子必须相互脱离, 此分离是由拓扑异构酶II作用完成的。这些酶具有断裂一条双链DNA分子并使第二条双链DNA分子通过此缺口的能力, 从而使这些连接的DNA分离。
后随链的合成不能复制线性染色体的最末端
end replication problem
末端复制问题(end replication problem): 当后随链复制机器到达染色体未端时, 有时引物酶没有足够的空间去合成新的RNA引物。这导致复制的不完整和后随链DNA产物上3’端形成一小段单链DNA。当此DNA产物在下轮被复制的时候, 两个产物中的一个将变短, 在上一轮复制中没有被复制的区域将丢失, 即末端复制问题。
解决线性染色体末端复制问题的办法:
- 一种解决方法是用蛋白质代替RNA作为每个染色体末端最后一个冈崎片段的引物。在这种情况下, “引物蛋白”与后随链模板结合并用一个氨基酸来提供-OH(通常为酪氨酸),以代替正常情况下RNA引物提供的3’-OH。通过与最后的后随链结合, 引物蛋白与染色体的3 ‘端形成共价连接。这种在末端连接复制蛋白质的情况, 在某些具线性染色体的细菌(多数细菌是环形染色体)的染色体末端, 以及在某些具线性染色体的细菌病毒和动物病毒的染色体末端都有发现。’
- 多数真核细胞使用完全不同的方法来复制其染色体末端。
真核生物染色体的末端称为端粒,它们通常由首尾相接的、富含TG的DNA序列构成。这种独特的结构可作为新的复制起始位点以弥补末端复制问题。此起始位点募集端粒酶(telomerase)。端粒酶为后随链复制机器的作用提供了额外的模板, 在后随链DNA复制机器的作用下, 使细胞也能有效地增加染色体5’端的长度。在后随链复制机器作用后, 染色体末端仍有一小段单链DNA区域——3’端凸出尾巴, 它的存在对端粒的末端保护功能很重要。端粒酶和后随链复制机器的作用, 能够保证端粒维持在足够的长度以保护染色体末端不会变得太短, 端粒DNA的重复性及非蛋白编码特性, 使细胞可以耐受不同长度的端粒存在。以此解决了线性染色体末端复制问题。
端粒酶是一种不需要外源模板的新型DNA聚合酶
端粒酶(telomerase): 是一种特殊DNA聚合酶, 它既含有多蛋白亚基, 也含有RNA成分, 端粒酶发挥特殊功能的关键所在是该酶的RNA组分。端粒酶的一个蛋白质亚属于DNA聚合酶, 称为反转录酶, 用于延伸其DNA底物的3’端, 不需要外源性DNA模板来指导dNTP的添加, 利用其RNA成分作为模板, 将端粒序列添加到染色体末端, 特异性地延伸特定单链DNA序列的3’-OH。
端粒酶RNA: 端粒酶发挥特殊功能的关键所在是该酶的RNA组分, 称为端粒酶RNA或TER,在不同物种中, TER大小从150到1300碱基不等。在所有生物中, 此RNA序列含有1.5拷贝的完整端粒序列,能与端粒3’端的单链DNA退火。
端粒酶与DNA聚合酶的异同之处:
端粒酶作为一种特殊的DNA聚合酶既有与之相同之处, 又存在自己独特之处。
- 1.相同之处:
- 1.二者都为DNA聚合酶, 都可以进行DNA合成;
- 2.也需要模板来指导核苷酸的添加, 使用同样的核苷酸前体;
- 3.只能延长DNA的3’端;
- 4.具有持续延伸能力, 每次与DNA底物结合后可添加很多重复序列。
- 2.不同之处:
- 端粒酶的特点: 含有RNA成分、不需要外源性模板, 以及能够使用单纯的单链DNA底物。此外, 端粒酶还具有将其RNA模板从DNA产物上去除的能力, 以重复进行模板指导下的合成, 因此端粒酶有RNA-DNA解旋酶活性。
端粒酶通过延伸染色体3’端解决了末端复制问题
端粒酶在5’端的延伸:
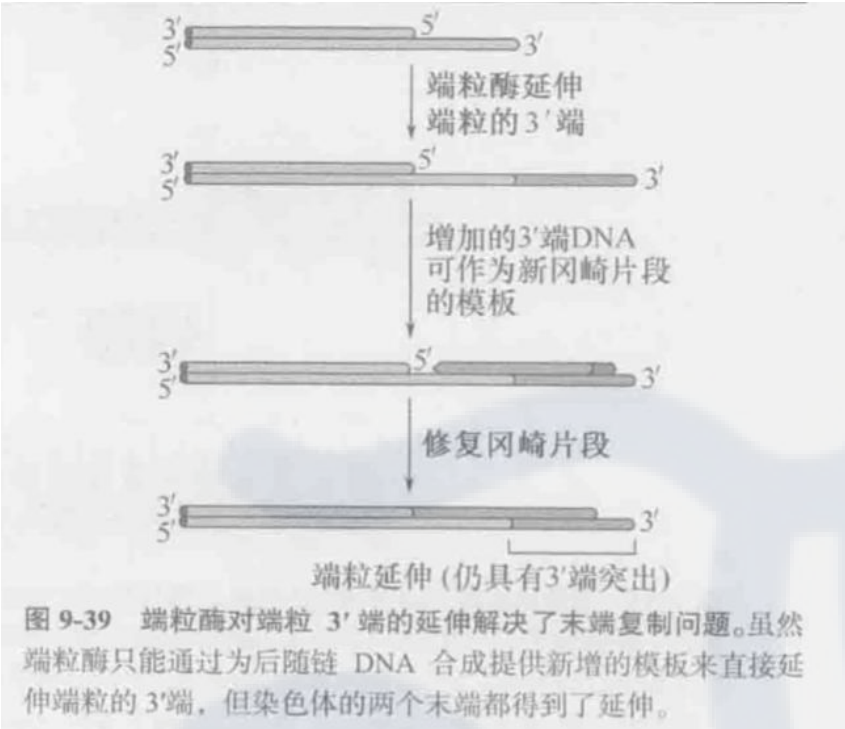
端粒结合蛋白调节端粒酶活性和端粒长度
如何维持端粒在所有染色体末端具有相同长度?
理论上,端粒酶可以无限制地延长端粒。但结合在端粒上的蛋白质是端粒酶活性的弱抑制剂。
当端粒序列重复拷贝较少时,较少的蛋白质与端粒结合,端粒酶可以延长端粒的3’羟基末端。
随着端粒的逐渐增长,更多的端粒结合蛋白聚积并且抑制端粒酶延长端粒的3”羟基末端。
这一简单的负反馈机制(长端粒抑制端粒酶)是维持端粒在所有染色体末端具有相同长度的强有力的方法。结合蛋白识别单链形式的端粒同样能调节端粒酶活性。”
端粒结合蛋白保护染色体末端
(功能见第八章)
什么在保护染色体末端?
- 端粒结合蛋白保护染色体末端至关重要。在细胞中, DNA末端的存在是DNA中双链断裂的标记, 该断裂是DNA修复机器的靶位点。这种修复最常见的结果是启动基因组上DNA之间的重组, 如果端粒参与该重组事件将会产生严重后果, 以修复双链DNA断裂的方式来修复端粒会导致染色体融合, 最终导致染色体随机断裂。结合在端粒上的蛋白质将端粒同细胞中其他DNA
- 对人类端粒结构的研究提供了另一种保护方法。电镜观察到从人类细胞中分离的端粒是环状结构而非线性结构,后续分析显示该结构(称为t环)是由端粒的3’单链DNA末端向端粒的双链DNA区域插入产生的。形成t环后端粒末端被包裹起来, 不能被识别为正常DNA。
考点补充
DNA复制相关酶/蛋白质
- 说出双链DNA复制其实有关的五种重要的酶或蛋白质, 并简述它们的功能。(2001)
- 蛋白质与核酸的相互作用存在于基因表达的各个水平上, 请分别举例说明在基因复制RNA转录和蛋白质翻译过程中二者的相互作用。(2005)
- 论述一个真核基因产生有功能的蛋白质需要经过哪些分子生物学过程?列出每个过程发生的细胞位置和参与的主要酶和蛋白质因子。(2006)
- 简述DNA复制中RNA引物的去除所需要的酶及其作用。(2012)
重要蛋白
DNA pol
SSB
DNA连接酶
引物酶
拓扑异构酶
解旋酶
复制子
复制器
起始子蛋白
引物的来源
由于DNA聚合酶没有从头合成DNA的功能, 在DNA复制起始阶段, 需要一种特殊的RNA聚合酶即引物酶在模板链附近合成引物, 通过引物提供3’-OH, DNA聚合酶才能够开始合成DNA。
要完成DNA的复制,用于起始的RNA引物就必须被除去并用DNA取而代之。对于去除引物, 不同生物通过类似但又有所不同的方式进行。
原核生物—DNA聚合酶I一步到位
大肠杆菌RNA引物的去除所需要的酶是DNA聚合酶I
DNA聚合酶I的作用
- DNA聚合酶I具有5’外切核酸酶的活性,能将DNA合成位点最上游的RNA或DNA迅速除去。
- 与DNA聚合酶Ⅲ不同,DNA聚合酶I的延伸能力不强, 每次结合仅能添加20~100个核苷酸。这些性质使其适用于RNA引物的去除和在所产生的单链DNA缺口中进行DNA的合成。DNA聚合酶I合成较短片段的能力使其适用于合成先前被RNA引物占据的短区域
- DNA聚合酶I的5’外切核酸酶可以除去RNase H所不能除去的RNA-DNA连接。
- DNA聚合酶I完成其功能后仅在DNA链上留一个缺口。这个缺口最后由DNA连接酶连接修复。
真核生物引物去除用到RNaseH和5’外切核酸酶
为了用DNA取代RNA引物, RNA酶H(RNase H)识别并除去各条RNA引物的大部分。此酶特异性地降解与DNA碱基配对的RNA(此处,其名称中的”H”意指RNA-DNA杂交链中的杂交,hybrid)。
RNase H可除去其他所有的RNA引物
不能去除DNA末端直接连接的核糖核苷酸。
这是因为RNase H只能断裂两个核糖核苷酸之间的键。
最后一个核糖核苷酸是由从5′端降解RNA或DNA的外切核酸酶除去的。
RNA引物除去后在双链DNA中留下的切口,形成引物-模板接头,这是DNA聚合酶理想的底物。
DNA聚合酶填补此切口直至每个核苷酸都碱基配对, 生成一条完整的DNA分子, 除了在磷酸二酯键骨架上被修复链的3’-OH和5′磷酸基之间有一处断裂。DNA上的这个”切口”被称为DNA连接酶(DNA ligase)的酶修复。DNA连接酶消耗ATP, 在相邻的5’磷酸基和3’-OH之间形成一个磷酸二酯键。只有当所有的RNA引物都被替代且相关的切口均被修复后, DNA的合成才告结束。)
共同利用了DNA连接酶
端粒与端粒酶
- 简述真核生物 DNA 复制中,端粒复制与染色体其他部分DNA复制的异同和生物学意义。(2004)
- 简述染色体端粒复制的问题,人正常细胞和癌细胞端粒复制有何不同?(2006)
- 2009 年三名美国科学家获诺贝尔生理学或医学奖,请分析他们为什么获奖,解释他们对分子生物学的贡献。并用他们的发现解释克隆羊多利早衰死亡的一个原因。(2010)
- 简述端粒酶在 DNA 复制中的功能及其生物学意义。(2014)
- 什么是端粒和端粒酶?有何功能?端粒最初在什么生物中发现?该生物有何优势?(2022简)
端粒的生物学意义
1.维持染色体稳定性(本身作用):
端粒酶能够维持染色体末端的端粒长度, 这对于染色体的稳定性和完整性至关重要。每次细胞分裂时, 染色体的端粒会缩短, 这是因为DNA聚合酶在复制DNA时无法完全复制染色体的末端。如果没有端粒酶, 端粒会逐渐缩短直到达到临界长度, 这会导致染色体融合、断裂或功能丧失, 引发基因组不稳定性和细胞衰老。
2.细胞分裂与寿命(对细胞的意义):
端粒酶在细胞中负责端粒的延长, 防止因细胞分裂而导致的端粒损耗, 这使得细胞能够进行更多的分裂周期而不致于过早衰老。在大多数体细胞中, 端粒酶的活性很低或不存在, 限制了细胞的分裂次数, 符合Hayflick界限, 即正常体细胞的有限分裂能力。
3.癌症与永生性(研究与癌细胞的关系):
在大多数恶性肿瘤细胞中, 端粒酶的活性异常升高, 这使得癌细胞能够无限分裂, 获得所谓的”永生性”。
4.干细胞与再生医学(研究与干细胞的关系):
干细胞和生殖细胞中通常有活跃的端粒酶, 这有助于保持这些细胞的自我更新能力和分化潜能。在再生医学领域, 了解端粒酶如何维持端粒长度对于开发细胞疗法和组织工程非常重要。
5.衰老与疾病(研究与衰老的关系):
端粒长度与衰老过程相关联, 短端粒与多种年龄相关性疾病如心血管疾病、神经退行性疾病和免疫功能下降有关。因此, 端粒酶和端粒长度的研究有助于理解衰老机制和寻找延缓衰老的策略。
端粒的发现
端粒最初在模式生物四膜虫中发现。它对于发现端粒和端粒酶的贡献就像线虫之于发见田胞凋亡一样。1978年, Liz女士利用这种特殊的模式生物纯化了rDNA, 以rDNA为模板通过体外合成参入的实验, 推断四膜虫的端粒是由许多重复的5’-CCCCAA-3’六个碱基序列组成的。
优势: 四膜虫有两个细胞核。小核很稳定, 含5对染色体, 用于生殖传代。而大核在接合细胞的发育过程中, 染色体断裂成200-300个小染色体, (含有编码核糖体RNA的基因)从染色体上断裂后通过复制更是形成高达10000个小染色体。四膜虫的小染色体众多, 也就说端粒可能非常丰富。这就为端粒研究提供了得天独厚的材料。
DNA解旋酶极性的实验验证
DNA复制的保真性
- DNA复制具有很高的准确性(accuracy)或保真度(fifelity), 出错率低于1010之一, 请解释: DNA复制过程中与复制后有哪些保证复制的正确性?为什么RNA聚合酶的保真度比DNA复制低?DNA复制的错误或突变对生物本身有何意义?(2007)
- 遗传信息在复制、转录和翻译过程中的准确性分别是如何实现的?(2008)
- DNA是如何保证复制过程中的精确性的?(2011)
- DNA复制过程中怎样保证精确性和完整性?(2016)
- DNA甲基化对DNA复制和修复的调控。(2022简)
回答两部分: 参与DNA复制的各个组分 + 复制结束后保证系统正确性的机制
补充: 为什么RNA聚合酶的保真度比DNA低?
1.校对功能的缺失:
DNA聚合酶通常具有3’→5’外切核酸酶活性, 这是一种校对机制, 能够在新合成的DNA链中检测并去除错配的核苷酸, 从而大大提高DNA复制的准确性。相比之下, RNA聚合酶缺乏这种3’→5’外切核酸酶活性, 它没有有效的校对机制来纠正转录过程中产生的错误, 因此转录的保真度较低。
2.模板和产物的稳定性:
DNA是双链结构, 提供了一套额外的检查机制, 因为两条链通过碱基互补配对相互验证。这增加了复制的准确性, 因为任何不匹配都会导致结构上的不稳定。RNA通常是单链结构, 没有这种双链的校验, 因此在合成过程中错误的核苷酸更容易被加入。
3.功能需求的差异:
DNA需要高度的保真度, 因为它携带着遗传信息, 必须在细胞分裂时准确无误地传递给后代细胞。RNA, 特别是mRNA, 主要作为蛋白质合成的模板, 虽然准确性很重要, 但它们的半衰期较短, 且通
常不需要像DNA那样精确的复制。
真核和原核DNA复制的差异
相同点
- 遗传方式相同, 都是半保留复制
- 办不连续性复制
- DNA合成方向为5’->3’
- 延伸过程类似, 所需材料也都为脱氧核糖核苷酸
- 需要引物和模板
- 具有高保证性
- 都需要多种酶和蛋白质协同参与, 都涉及拓扑异构酶, 解旋酶, DNA聚合酶以及连接酶
不同点
- 1.时期
- 1.1 真核: 分裂间期, S期
- 1.2 原核: 整个细胞周期
- 2.起点数目
- 2.1 真核: 每条染色体上可以多个
- 2.2 原核: 每个染色体上只有一个
- 3.引物
- 3.1 真核: 由DNA pol $\alpha$/引物酶合成
- 3.2 原核: 引物酶合成
- 4.主要的延伸酶
- 4.1 真核: 前导链和后随连各有一个
- 4.2 原核: DNA pol III
- 5.复制叉的移动速度
- 5.1 真核: 块
- 5.2 原核: 慢
- 6.冈崎片段的长度
- 6.1 真核: 100~400bp
- 6.2 原核: 1000~2000bp
- 7.解旋酶的装载
- 7.1 真核: G1期, DNA解旋, 两个解旋酶头对头包裹双链DNA
- 7.2 原核: 起始微店DNA发生部分解链, 装载在单链DNA上
- 8.pol的募集
- 8.1 真核: 在引物合成之前
- 8.2 原核: 由引物-模板接头吸引
- 9.起始调控机制
- 9.1 真核: CDK水平控制解旋酶装载激活
- 9.2 原核: 起始蛋白识别起始位点DNA
- 10.DNA复制与细胞周期是否欧联
- 10.1 真核: 欧联, 一次细胞周期只发生一次
- 10.2 原核: 不相关, 在快生长模式下, 正在合成中的DNA脸上可以开启新一轮复制
起始字蛋白
功能:
- 这些蛋白质与复制器中的特异DNA序列结合;
- 一旦与DNA结合, 它们就扭曲或解旋其结合位点附近的DNA区域:
- 起始子蛋白与复制起始所需的其他因子相互作用, 使它们聚集到复制器上。
复制与转录的差异
提高DNA结合酶衍生性的因素
- DNA聚合酶在复制叉上具有高延伸能力的关键原因在于它们与被称为滑动DNA夹的蛋白质间的结合。在无滑动夹的情况下,DNA聚合酶平均每合成20~100个碱基对就从DNA模板上解离并散落开来。在有滑动夹的情况下,DNA聚合酶的活性部位仍经常从DNA的3’-OH末端脱落,但是其与滑动夹的结合阻止了聚合酶从DNA上散开。通过保持DNA聚合酶与DNA的紧密接触,滑动夹使DNA聚合酶能迅速地重新结合到同一引物模板接头上,大大增加了DNA聚合酶的延伸能力。
- DNA聚合酶拇指域与最新合成的DNA相互作用,维持引物和活性位点的正确位置,帮助维持DNA聚合酶与其底物之间的较强的结合,使DNA聚合酶每次与引物-模板接头结合时,具有添加多个dNTP的能力,因此提高了DNA聚合酶的延伸能力。
- DNA聚合酶沿DNA滑行的能力有利于提高延伸能力,其中有两种利于DNA聚合酶沿DNA滑行的相互作用:
- 拇指域和磷酸骨架间静电作用(序列非特异性)
- 手掌域与小沟间的氢键作用(序列非特异性,每添加一个核苷酸断裂一次)
名词补充
Telemerase端粒酶: 是一种特殊DNA聚合酶, 它既含有多蛋白亚基, 也含有RNA成分, 端粒酶发挥特殊功能的关键所在是该酶的RNA组分。端粒酶的一个蛋白质亚属于DNA聚合酶, 称为反转录酶, 用于延伸其DNA底物的3’端, 不需要外源性DNA模板来指导dNTP的添加, 利用其RNA成分作为模板, 将端粒序列添加到染色体末端, 特异性地延伸特定单链DNA序列的3’-OH。
RNase H
RNA酶H(RNase H): 能够识别并除去各条RNA引物的大部分。RNase H只能断裂两个核糖核苷酸之间的键, 因此除了与DNA末端直接连的核糖核苷酸之外, 此酶能特异性降解与DNA碱基配对的RNA。