分子生物学 第十五章 翻译后修饰
1
2
3
4
5
6
7
8
9 ______ ___ __
/\__ _\ /\_ \ /\ \__ __
\/_/\ \/ _ __ __ ___ ____\//\ \ __ \ \ ,_\/\_\ ___ ___
\ \ \/\`'__\/'__`\ /' _ `\ /',__\ \ \ \ /'__`\ \ \ \/\/\ \ / __`\ /' _ `\
\ \ \ \ \//\ \L\.\_/\ \/\ \/\__, `\ \_\ \_/\ \L\.\_\ \ \_\ \ \/\ \L\ \/\ \/\ \
\ \_\ \_\\ \__/.\_\ \_\ \_\/\____/ /\____\ \__/.\_\\ \__\\ \_\ \____/\ \_\ \_\
\/_/\/_/ \/__/\/_/\/_/\/_/\/___/ \/____/\/__/\/_/ \/__/ \/_/\/___/ \/_/\/_/
本章概要
翻译(translation): 指将mRNA上的核苷酸从一个特定的起始位点开始, 按每3个核苷酸代表一个氨基酸的原则, 依次合成一条多肽链, 将蕴藏在mRNA核苷酸序列中的遗传信息被解读并产生蛋白质氨基酸序列这一过程称作翻译。
翻译过程中面临的主要挑战:
- mRNA中的遗传信息不能被氨基酸识别;
- 遗传密码必须被接头分子识别, 接头分子必须准确招募氨基酸。
翻译机器:
- 信使RNA
- 转运RNA
- 氨酰tRNA合成酶
- 核糖体
mRNA的蛋白质编码区是由称为密码子(codon)的一系列有序的三核苷酸单位组成的, 密码子的排列顺序将决定氨基酸的排列顺序。
信使RNA
多肽链是由可读框规定的
翻译机器只解码每条mRNA的部分序列。蛋白质合成的信息蕴藏在三核苷酸密码子中, 每个密码子指定一个氨基酸。
可读框(open-reading frame, ORF): 又称开放阅读框, 指的是从起始密码子开始, mRNA序列中具有编码蛋白质潜能的序列, 到终止密码子结束的连续碱基序列。真核细胞的mRNA几乎都只有一个ORF, 而原核细胞的mRNA经常含有两个或多个ORF。
起始密码子(start codon): 可读框第一个密码子称为起始密码子。细菌中, 起始密码子通常是5’-AUG-3’, 但是5’-GUG-3’有时甚至5’-UUG-3’也使用。真核细胞总是使用5’-AUG-3’为起始密码子。起始密码子有两个重要的功能: 第一, 它指定掺入到增长的多肽链中的第一个氨基酸;第二, 它确定了所有后续密码子的可读框。
终止密码子(stop codon): 可读框最后一个密码子称作终止密码子。无论是真核还是原核细胞, 终止密码子有三个(5’-UAG-3’、5’-UGA-3’、5’-UAA-3’),它们决定可读框的终点并发出终止多肽合成的信号。
信使RNA包含至少一个可读框。每条mRNA的ORF数量在真核和原核细胞中的区别: 原核细胞的mRNA经常含有两个或多个ORF,因此可编码多个多肽链;真核细胞的mRNA几乎都只有一个ORF。
多顺反子mRNA(polycistronic mRNA): 含有多个ORF的信使RNA称为多顺反子mRNA。
单顺反子mRNA(monocistronic mRNA): 只编码一个ORF的信使RNA称为单顺反子mRNA。
真核与原核mRNA的区别:
- 原核mRNA具有多个开放阅读框, 真核mRNA只有单个开放阅读框;
- 原核一个mRNA产生多个多肽产物, 真核只产生1个多肽产物;
- 原核细胞mRNA没有5’帽子和3’尾巴, 真核细胞mRNA有;
- 原核细胞mRNA有RBS, 真核细胞没有。
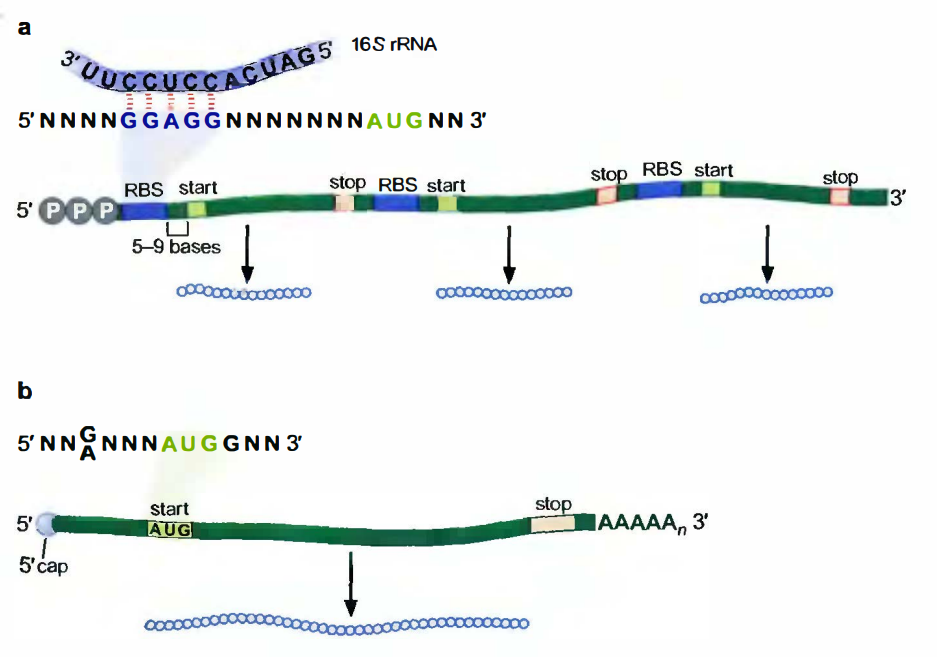
原核细胞mRNA具有核糖体结合位点,可以募集翻译机器
核糖体结合位点(ribosome binding site, RBS)或SD序列(Shine-Dalgarno sequence): 位于许多原核细胞的可读框在起始密码子的上游(5’端)3-9nt含有一段短序列称为核糖体结合位点, 这一元件也被称为SD序列。原核细胞的RBS(GGAGG)序列与核糖体RNA组分之一的16S核糖体RNA的3’端的一段序列(CCUCC)互补配对, 使得核糖体招募在可读框的起始处, 起始翻译。
翻译耦合(translational coupling): 5’-AUGA-3’序列绕开了用RBS募集核糖体的需要, 刚刚结束上游可读框翻译的核糖体正好处于恰当的位置, 并从下游可读框的起始密码子开始翻译, 这种交叠可读框之间的连锁翻译称为翻译耦合。下游ORF的翻译的进行需要依赖于上游ORF的翻译, 上游ORF中的终止突变也会阻止下游ORF的翻译。
真核细胞mRNA在5’和3’端被修饰,以促进翻译
真核细胞的mRNA通过位于5’端的称为5’帽子(5’cap)的特殊化学修饰来募集核糖体。
扫描(scanning): 形成的5’帽子结构募集核糖体结合到mRNA上, 一旦结合到mRNA上, 核糖体沿着5’→3’的方向移动直至遇到5’-AUG-3起始密码子, 这一过程称为扫描。
真核生物mRNA的两个特征能促进翻译过程:
- 其一是在某些mRNA中, 起始密码子的上游第三个碱基为嘌呤并且第一个下游碱基为鸟嘌呤的特殊序列(5’-G/ANNAUGG-3’)。这一序列称为Kozak序列。Kozak序列被认为能够与起始tRNA发生相互作用, 而不是与核糖体的RNA成分相互作用。
- 另一个有利于翻译的特征是3’端的poly-A尾结构。poly-A尾巴可通过增强关键翻译起始因子的募集作用来提高翻译的水平。3’端的修饰也可以保护真核mRNA免受快速降解。
转运RNA
tRNA是密码子和氨基酸之间的转配器
蛋白质合成的核心是将核苷酸序列的信息(以密码子的形式)”翻译”成氨基酸。这是由tRNA分子完成的, 它担当密码子及其所决定的氨基酸之间的转配器。
转运RNA(tRNAs): 分子长度为75-95nt, tRNA分子有很多种, 每种仅与一个特定的氨基酸结合, 每种识别mRNA的一个或几个特定的密码子, 所有tRNA末端均为5’-CCA-3’序列, 其3’端是氨酰tRNA合成酶加载氨基酸的位点。
tRNA的种类:
- 起始tRNA和延伸tRNA: 能特异性识别mRNA模板上起始密码子的tRNA叫起始tRNA。其它的统称延伸tRNA。
- 同工tRNA: 代表同一种氨基酸的tRNA为同工tRNA。
- 校正tRNA: 分为无义突变和错义突变校正tRNA, 无义突变的校正tRNA可改变反密码子区校正无义突变。错义突变的校正tRNA通过反密码子区的改变把正确的氨基酸加到肽链上, 合成正常的蛋白质。
tRNA一级结构中存在特殊碱基:
- 尿嘧啶、假尿嘧啶ψU、双氢尿嘧啶DU;
- 次黄嘌呤、胸腺嘧啶、甲基鸟嘌呤, 这些修饰碱基对tRNA的功能并非不可或缺, 但缺少这些碱基的细胞生长变慢。这一现象暗示这些修饰碱基可能可以提高tRNA的功能。
tRNA具有共同的三叶草型的二级结构
tRNA分子表现为特异的, 高度保守的单链和双链区域(二二级结构),该结构可描绘成三叶草型。tRNA三叶草型结构的主要特征是具有1个受体臂、3个茎环(分别为PU环、D环和反密码子环)和1个可变环。
- 受体臂(acceptor stem): 由于具有结合氨基酸的位点而得名, 由tRNA分子的5’和3’端的碱基配对而成。其3’端的5’-CCA-3’序列突出双链之外。
- PU环(PU loop): 因特殊碱基PU(假尿嘧啶)的存在而得名。这一修饰碱基经常发现位于5’-TYUCG-3’序列中。
- D环(D loop): 因特殊碱基双氢尿嘧啶的存在而得名。
- 反密码子环(anticodon loop): 包含反密码子——一个三核苷酸序列, 能够通过碱基配对方式识别mRNA的密码子。反密码子的两端由5’端的尿嘧啶和3端的嘌呤界定。
- 可变环(variable loop): 位于反密码子环和PU环之间, 长度为3~21bp不等, 正如其名。
tRNA具有L形的三维结构
在最终形成的tRNA三维构象中, 受体臂和PU环茎之间将形成延伸的螺旋结构。同样地, 反密码子茎和D环茎形成第二个延伸的螺旋。这两个延伸的螺旋垂直排列, D环和PU环靠在一起。
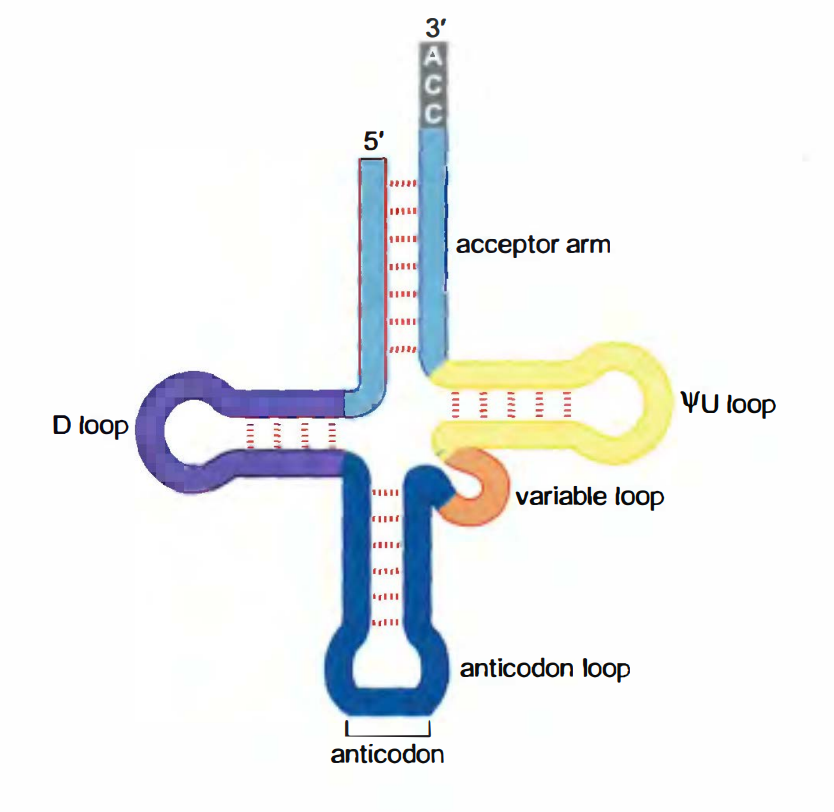
三种相互作用使L状结构得以稳定:
- 三维结构中相互靠近的不同螺旋区域的碱基间的氢键, 这些通常是非常规(非Watson-Crick)键;
- 碱基和磷酸核糖骨架之间的相互作用;
- 因形成两个延伸的碱基配对区域而获得的碱基堆积作用。
氨基酸连接到tRNA上
氨基酸通过高能酰基连接到tRNA 3’端的腺苷酸上使tRNA负载
负载tRNA(charged tRNA): 连接了氨基酸的tRNA分子称为负载tRNA。
空载RNA(uncharged tRNA): 未连接氨基酸的tRNA称为空载tRNA。
负载过程需要氨基酸的羧基与tRNA 3’端受体臂突出的腺苷酸的2’或3’羟基一个高能键, 高能键水解所释放的能量帮助推动多肽链中氨基酸之间肽键的形成。
氨基酰tRNA合成酶分两步使tRNA负载
tRNA负载过程:
- 第一步是氨基酸的腺苷酰基化(adenylylation)。氨基酸和ATP反应而被腺苷酸酰基化, 结果AMP的磷酸基团与氨基酸的羧基反应形成高能键释放出焦磷酸, 腺苷酰基化的氨基酸与氨酰tRNA合成酶紧密结合;
- 第二步是tRNA负载, 酰基化的氨基酸与tRNA反应, 结果, 酰基化的氨基酸通过2’或3’羟基被转移到tRNA的3’端, 同时释放出AMP。
tRNA合成酶分两类:
- Ⅰ类酶将氨基酸连接到tRNA的2’羟基, 并且通常是单聚体;
- Ⅱ类酶将氨基酸连接到tRNA的3’羟基, 并且通常是二聚体或四聚体。
每种氨基酰tRNA合成酶连接一个氨基酸到一个或多个tRNA上
20种氨基酸的每一种都是通过单独专门的tRNA合成酶连接到正确的tRNA上的。由于大多数的氨基酸是由多于一个的密码子决定的, 一个合成酶识别多于一个tRNA。这些tRNA统称为同工tRNA。
对于一个氨基酸而言只有一种tRNA合成酶将每个氨基酸连接到所有正确的tRNA上。大多数的生物具有20种不同的tRNA合成酶。
氨基酸-反密码子-密码子-tRNA-tRNA合成酶的关系:
一个氨基酸-多个密码子-多个反密码子-多个tRNA-一个tRNA合成酶
大多数的生物具有20种不同的tRNA合成酶, 但并不总是这样。例如, 有些细菌缺乏使谷酰胺连接到$tRNA^{GIn}$的合成酶。取而代之的, 是由一种氨酰-tRNA合成酶负责将Glu连接到$tRNA^{Gln}$和$tRNA^{Glu}$上。然后第二二种酶将一部分负载的$tRNA^{Gln}$上的Glu氨基化形成Gln。即将$Glu-tRNA^{Gln}$氨基化变成$GIn-tRNA^{Gln}$(前缀指氨基酸, 上标指tRNA所识别的密码子)。第二种酶的存在使细菌不再需要谷酰胺tRNA合成酶。但是, 一种氨酰tRNA合成酶永远不能将多于一种的氨基酸连接到-种其相应的tRNA上。
tRNA合成酶识别同族tRNA(cognate tRNA)的独特结构
氨酰-tRNA合成酶必须保证两个水平的准确性:
- 它们必须识别针对特定氨基酸的一组正确的tRNA;
- 它们必须使这些同工tRNA负载正确的氨基酸。
tRNA识别的特异性(tRNA合成酶靠什么识别同工tRNA):
特异性的决定因素集中在tRNA分子的两个不同的位点: 受体臂和反密码子环。
- 受体臂: 是tRNA合成酶特异性识别的一个极其重要的决定因素。在某些情况下, 改变受体臂的一个碱基[称为鉴别者碱基(discriminatorbase)]足以使特异性识别某个tRNA的合成酶变为另一种合成酶。
- 反密码子环: 经常对识别特异性有所贡献。由于一个氨基酸常由一个以上密码子编码, 在很多情况下对反密码子的识别并不起作用。
第二遗传密码(secondary code):tRNA分子上使氨酰tRNA合成酶特异性识别不同tRNA的一组决定结构, 称为第二遗传密码, 远比第一遗传密码复杂, 不易列成表格, 在基因信息的传递中起重要作用。
氨基酰tRNA的形成时非常精确的
提高氨基酰tRNA合成酶保真度的一个普遍机制:
利用编辑口袋来对负载反应的产物进行校对, 其作用与DNA聚合酶的校对识别机制类似。
核糖体
核糖体(ribosome): 是指导蛋白质合成的大分子机器。
核糖体是由一个大亚基和一个小亚基组成的
核糖体是由RNA和蛋白质组成的大亚基和小亚基两个部件组成的。
大亚基含有肽基转移酶中心(peptidyl transferase center), 负责肽键的形成。
小亚基含有解码中心(decoding center), 负载氨基酸的tRNA在此阅读或”解码”mRNA的密码子单位。
按照惯例,大、小亚基的命名是根据离心时的沉降速率而定的。测量沉降速率的单位是Svedberg(S;S值越大,沉降速率越快,分子越大)。
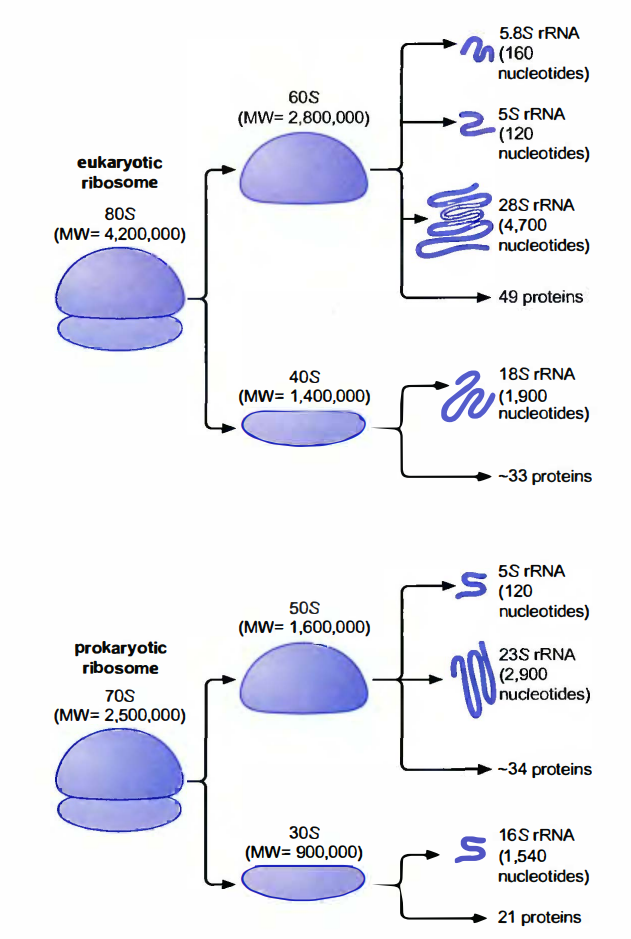
大小亚基在每一循环周期的翻译过程中都经历结合与分离
核糖体循环(ribosome cycle): 蛋白质每合成一次, 翻译机器就经历一系列特定的事件:核糖体的大、小亚基相互之间结合后与mRNA相结合, 以翻译目标mRNA, 完成蛋白质合成后则相互分离, 这种结合和分离相间的顺序称为核糖体循环。
多聚核糖体(polyribosome或polysome): 在蛋白质合成过程中,同一条mRNA分子能够同多个核糖体结合, 同时合成若干条蛋白质多肽链, 结合在同一条mRNA上的核糖体就称为多聚核糖体。多个核糖体和单个mRNA分子的结合方式确保在任何时候大多数的核糖体都能参与翻译。
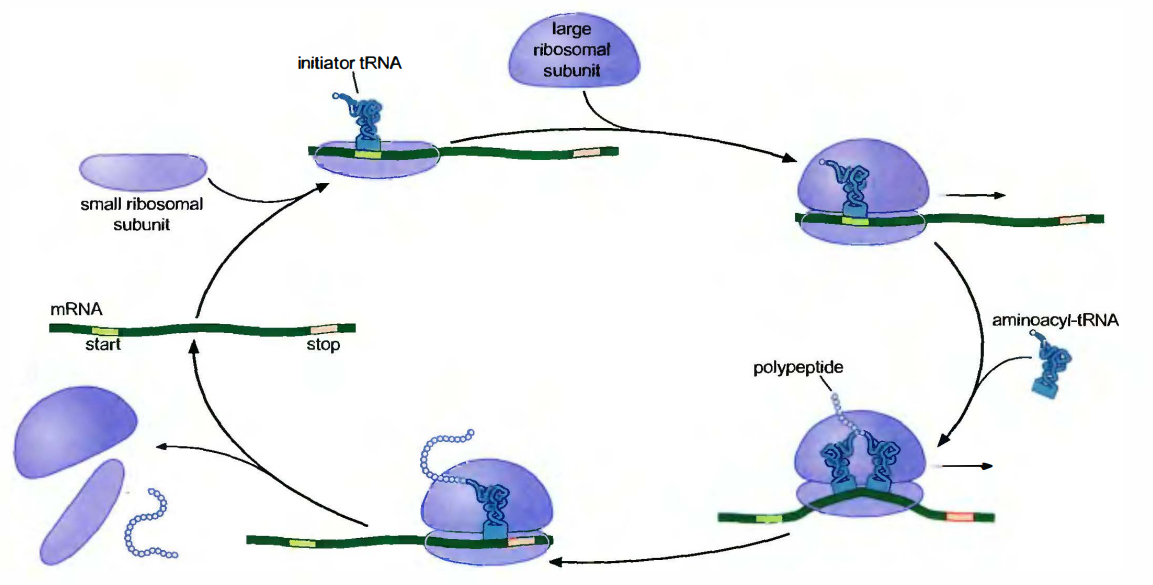
新的氨基酸连接在延伸肽链的碳末端
肽链合成方向:氨基端 -> 羧基端
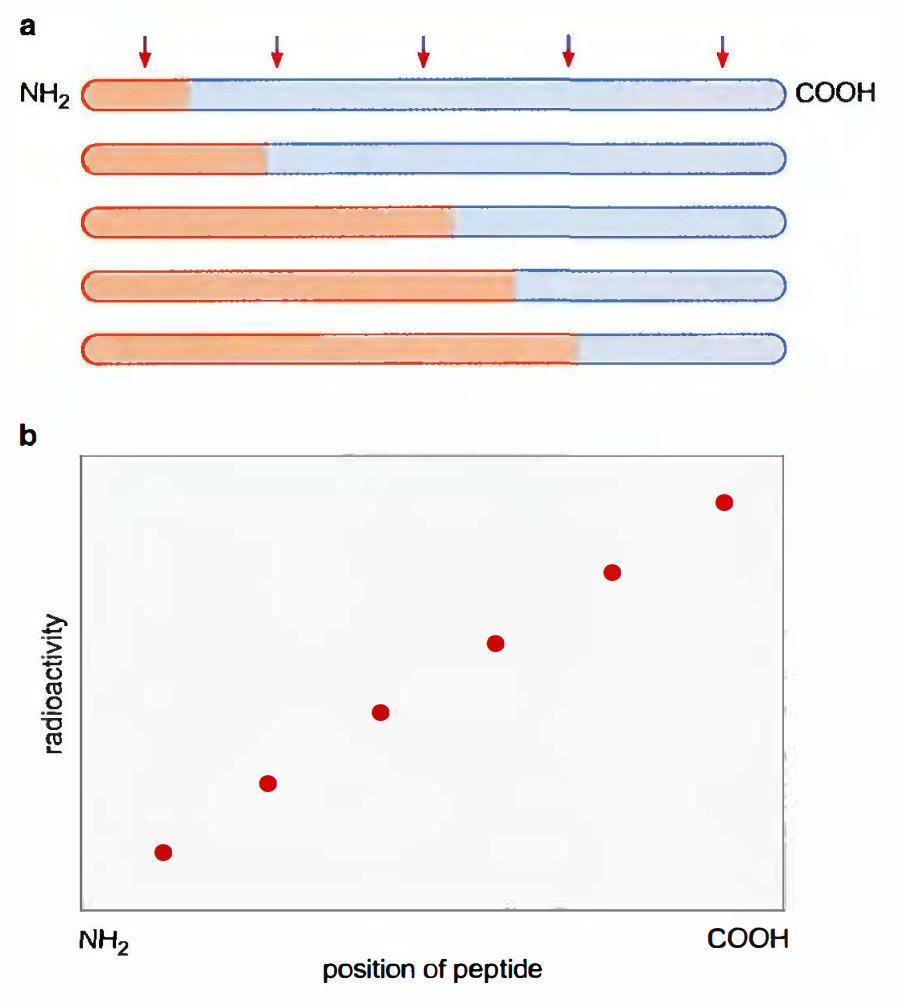
实验验证:
The nature of the genetic code, once determined, led to further questions about how a polynucleotide chain directs the synthesis of a
polypeptide. As we have seen here and shall discuss in more detail in
Chapter 6, polynucleotide chains (both DNA and RNA) are synthesized in a 5 ‘ —-+ 3’ direction. But what about the growing polypeptide
chain? Is it assembled in an amino-terminal to carboxyl-terminal
direction, or the opposite?
This question was answered in a classic experiment in which a
cell-free system was used for carrying out protein synthesis. The
cell-free system was created using an extract from immature red
blood cells (known as reticulocytes) from a rabbit, which are efficient factories for the synthesis of the a- and �-globin subunits of
hemoglobin. The cell-free system was treated with a radioactive
amino acid for a very few seconds (less than the time required to
synthesize a complete globin chain) after which protein synthesis
was immediately stopped. A brief radioactive labeling regime of this
kind is known as a pulse or pulse-labeling. Next, globin chains that
had completed their growth during the period of the pulse-labeling
were separated from incomplete chains by gel electrophoresis
(Chapter 20). The full-length polypeptides were then treated with an
enzyme, the protease trypsin, that cleaves proteins on particular
sites in the polypetide chain, thereby generating a series of peptide
fragements. In the final step of the experiment, the amount of
radioactivity that had been incorporated into each peptide fragment
was measured (Figure).
Keep in mind that the globin chains were at various stages of
completion during the period of the pulse (Figure 2-19a). Thus,
nascent chains that had only just started to be synthesized would
be unlikely to have reached completion during the period of the
pulse because the time of the pulse-labeling was less than the time
required to synthesize a complete globin chain. On the other hand,
globin chains that were almost full length would be highly likely to
have reached completion during the pulse. Also, keep in mind that
only chains that had reached full length during the time of the pulse
were isolated and subjected to trypsin treatment. It, therefore, follows that the trypsin-generated peptides with the least amount of
radioactive amino acid (normalized to the size of the peptide)
should have derived from regions of the globin protein that were the
first to be synthesized. Conversely, peptides with the greatest
amount of radioactivity should have derived from regions of the
protein that were the last to be synthesized.
The results of the experiment are shown in Figure. As you
can see, radioactive labeling was lowest for peptides from the aminoterminal region of globin and greatest for peptides from the carboxylterminal region. We, therefore, conclude that the direction of protein
synthesis is from the amino-terminus to the carboxyl-terminus. In
other words, during protein synthesis the first amino acid to be
incorporated into the nascent chain is the amino acid at the amino
terminal end of the protein and the last to be incorporated is at the
carboxyl-terminus.
肽键是通过将正在延伸的多肽链从一个tRNA转移到另一个tRNA而形成的
肽键是通过将正在延伸的多肽链从一个tRNA转移到另一个tRNA而形成的, 多肽合成的这一方式产生了两个后果:
- 这个肽键形成的原理要求蛋白质的氮端先于碳端合成;
- 多肽链从肽酰-tRNA转移到氨酰-tRNA上。
因此, 新肽键形成的反应称为肽酰转移酶反应(peptidyl transferase reaction)。
肽键的形成并没有伴随三磷酸核苷的水解。是因为肽键的形成是由连接多肽链和tRNA的高能酰键的断裂来驱动的。这一高能酰键是在tRNA负载氨基酸时由tRNA合成酶催化产生的。tRNA负载氨基酸的反应伴有一个ATP分子的水解。因此肽键形成所需的能量来自于tRNA负载氨基酸时所水解的那个ATP分子。
核糖体RNA是核糖体中的结构和催化决定因子
核糖体RNA并不单单是核糖体的结构成分, 相反, 它们还直接负责核糖体的关键功能。大多数的核糖体蛋白质位于核糖体的周边, 而非其内部。
核糖体有3个tRNA结合位点
核糖体含有3个tRNA结合位点, 分别为A、P和E位点。A位点(A site)是氨基酰tRNA的结合位点, P位点(P site)是肽基酰tRNA的结合位点, E位点(E site)是延伸的多肽链转移到氨基酰-tRNA后释放的tRNA的结合位点(E指exit, 退出)。
穿过核糖体的通道可以使mRNA和多肽链进出核糖体
核糖体通道及作用:
mRNA通过小亚基的两个狭窄的通道进出解码中心。mRNA的两个密码子之间存在明显的扭结(kink), 有利于维持正确的可读框。这个扭结使核糖体易位后留在空着的A位点的密码子处于一个独特的位置, 阻止随后的氨基酰-tRNA结合于毗邻该密码子的碱基上。
大亚基上的另一个通道提供了新合成的多肽链离开(核糖体)的途径。与mRNA的通道一样, 这个通道的大小限制了多肽链的折叠。
翻译的起始
原核细胞的mRNA最初是通过与rRNA的碱基配对而募集到小亚基上
翻译要成功地起始的3个条件:
- 核糖体必须被募集到mRNA上;
- 负载tRNA必须置于核糖体的P位点;
- 核糖体必须精确地定位在起始密码子上。其中第三条是关键的。因为这一步确定了mRNA翻译的可读框。
负载着修饰甲硫氨酸的tRNA直接结合到原核细胞的小亚基上
起始tRNA(initiator tRNA): 翻译起始过程一个特定tRNA, 它与起始密码子(通常AUG或GUG)碱基配对。
通常, 负载tRNA进入核糖体的A位点并在一轮肽键合成后才能到达P位点。但是, 在起始阶段, 负载tRNA直接进入到P位点。
起始tRNA上负载的既非Met亦非Val。相反, 它负载的是一种Met的修饰产物: 在氨基基团上连着一个甲酸基(N-甲酰甲硫氨酸, N-formyl methionine), 负载此氨基酸的起始tRNA称为$fMet-tRNA_i^{fMet}$。
所有原核生物的蛋白质的氨基端都有甲酰基团?
实际上, 许多原核细胞的蛋白质甚至不是以Met开始的, 因为去甲酰化酶(deformylase)能够在多肽链合成的进行过程中或合成过程之后将这个甲酰基从氨基端去除掉;氨肽酶(aminopeptidase)通常在氨基端切除Met及另外一两个氨基酸。
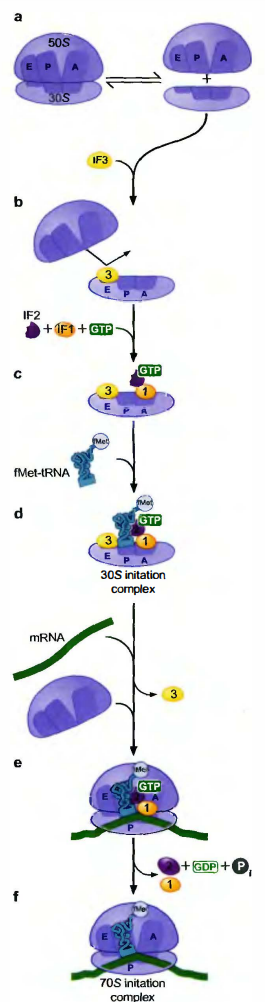
原核细胞翻译起始过程
原核细胞的翻译始于小亚基, 由三种翻译起始因子(translation initiation factor,IF)——IF1、IF2和IF3所催化。
每一个因子催化起始过程的一个关键步骤:
- IF1防止tRNA结合到小亚基中将形成A位点的位置上。
- IF2是GTP酶(结合和水解GTP的蛋白质), 它与起始过程的3个主要成分(小亚基、IF1和负载起始tRNA,即fMet-tRNAifMet)相互作用。通过与这些成分相互作用, IF2促使fMet-tRNAifMet和小亚基的结合, 并阻止其他负载tRNA与小亚基结合。
- IF3与小亚基结合并阻止其与大亚基重新结合。由于翻译起始需要游离的小亚基, 因此IF3与小亚基的结合对于翻译的新一轮循环是十分重要的。IF3在上一轮循环即将结束时开始与小亚基结合, 并协助70S核糖体分解为大、小亚基。
起始:
- 每一个起始因子能够结合到小亚基中的三个tRNA结合位点中的其中一个或其附近。为防止负载tRNA结合到A位点, IF1能够直接结合到小亚基中将形成A位点的位置上。IF2结合IF1并横跨A位点和P位点以结合fMet-tRNAMet。最后, IF3占据了将成为E位点的小亚基上的位置。这样, 在小亚基的三个潜在的tRNA结合位点中, 只有P位点在起始因子存在的条件下能够结合tRNA。
- 随着三个起始因子的加入, 小亚基已经准备好结合mRNA和起始tRNA。这两种RNA能以任何一种方向结合, 并且互相独立。小亚基结合mRNA需要mRNA的RBS和小亚基的16S RNA之间的碱基配对。而fMet-tRNAifMet结合小亚基过程的促进是通过与结合GTP的IF2相互作用, 并且(一旦mRNA结合于小亚基上)由反密码子和mRNA的起始密码子之间的碱基配对作用来实现的。类似地, fMet-tRNAifMet和mRNA之间的碱基配对起到了定位起始密码子到P位点上的作用。
- 翻译起始过程的最后步骤涉及大亚基的聚合以形成70S起始复合体(70S initiation complex)。当起始密码子和fMet-tRNAifMet的碱基配对后, 小亚基的构象发生变化, 导致IF3的释放。在IF3离开的情况下, 大亚基可以自由地与小亚基及其负载的IF1、IF2、mRNA和fMet-tRNAifMet结合。而且, IF2可以作为大亚基的初始对接位点, 并且这种相互作用激活了IF2-GTP的GTP酶活性。因此, IF2-GDP与核糖体和起始tRNA的亲和力降低, 导致IF2-GDP和IF1从核糖体释放出来。这样, 翻译起始过程的最终产物是在mRNA的起始位点组装了一个完整的70S核糖体, 其P位点有fMet-tRNAifMet,而A位点是空的。这个核糖体-mRNA的复合体已准备好在A位点结合负载tRNA,开始多肽链的合成。
真核细胞的mRNA通过5’帽吸引核糖体
真核细胞翻译的起始过程与原核细胞相同之处:
它们都使用起始密码子和专门的起始tRNA,都在大亚基的加入之前利用起始因子形成结合mRNA的小亚基复合体。
真核细胞翻译的起始过程与原核细胞不同之处:
真核细胞运用一个与原核细胞根本不同的方式来识别mRNA和起始密码子。
在真核细胞中, 小亚基在被募集到mRNA的5’端帽子结构之前, 已经与起始tRNA结合。然后以5’→3方向沿着mRNA”扫描”直到遇见第一个5’-AUG-3’,该密码子被识别为起始密码子。因此, 在大多数情况下,只有第一个AUG才能作为真核细胞翻译的起点。
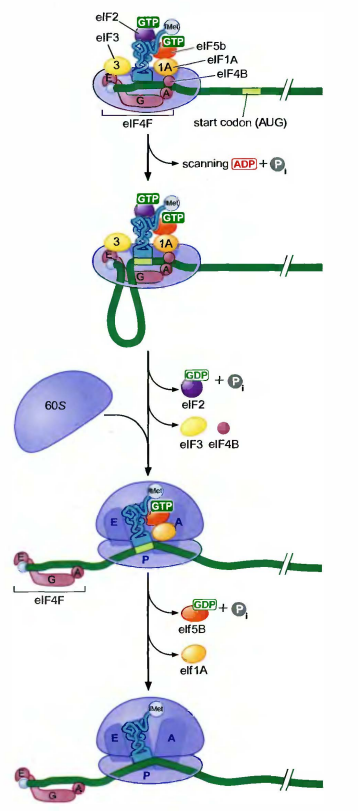
真核细胞翻译起始过程:
- 与在原核生物中不同, 在真核细胞中起始tRNA与核糖体小亚基的结合总是先于其与mRNA的结合。
- 由另一组不同的辅助因子介导对mRNA的识别。
- 结合于起始tRNA的核糖体在mRNA上”扫描”以搜寻第一个AUG序列。
- 核糖体的大亚基在起始tRNA与起始密码子碱基配对之后被募集进来。
当真核细胞的核糖体完成翻译的一个循环后, 通过与原核的IF3和IF1相对应的(分别称为eIF3和eIF1A)因子的作用解离为游离的大、小亚基。两个GTP-结合蛋白——eIF2和eIF5B介导了(小亚基)和负载起始tRNA的结合。
对真核细胞来说, 起始tRNA负载的是Met, 而非N-fMet, 称为Met-tRNAiMet。eIF5B-GTP帮助eIF2-GTP和Met-tRNAiMet的复合体结合到小亚基上。通过协同作用, 这两个GTP结合蛋白将Met-tRNAiMet安置于小亚基未来的P位点, 最后形成43S起始前复合体(43S pre-initiation complex)。43S起始前复合体对mRNA识别机制:
43S起始前复合体对mRNA的识别是从对5’帽结构的识别开始的, 这一结构存在于大多数真核细胞的mRNA上。这一识别过程由具有3个亚基的eIF4F介导。其中的一个亚基直接与5’帽结构相结合, 其他的两个亚基与RNA非特异性地结合。随后, eIF4B加入到这一复合体上, 并激活eIF4F中一个亚基的RNA解旋酶的活性。解旋酶解开mRNA末端的任何二级结构(如发夹结构)。eIF4F/B与展开的mRNA的结合体通过eIF4F和eIF3的相互作用募集43S起始前复合体到mRNA上, 形成48S前起始复合物(48Spre-initiationcomplex)。

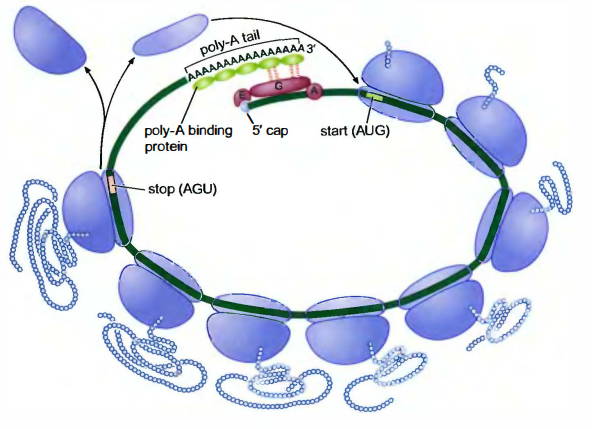
翻译起始因子使真核mRNA保持环状
除了与mRNA的5’端结合, 起始因子也可以通过poly-A尾与mRNA的3’端紧密结合。这一相互作用主要通过eIF4G和mRNA3’末端的直接结合以及与包绕在poly-A尾的poly-A结合蛋白(poly-A bindingprotein)的结合作用实现的。这种作用使5’端和3’端之间的分子紧密相邻, 并且使mRNA维持一种环状的构象。与polyA尾结构能够提高mRNA翻译效率的情况相似, 这些相互作用, 包括elF4E结合mRNA帽子和大亚基的募集过程, 能够提高翻译起始过程中某些步骤的效率。
起始密码子是通过核糖体从mRNA的5’端向下游(3’端)“扫描”而找到的
一旦在mRNA的5’端组装好, 小亚基和它的结合因子就按照5’→3’方向沿着mRNA移动, 这一移动是依赖于ATP的, 由eIF4F的RNA解旋酶所驱动。在移动过程中, 小亚基寻找mRNA的起始密码子。起始密码子的识别是通过起始tRNA的反密码子和起始密码子之间的碱基配对作用(这就是为什么起始tRNA先与小亚基结合, 而后与mRNA结合的原因)。正确的碱基配对引起eIF2和eIF3的释放。eIF2(防止大亚基的结合)和eIF3(结合于起始tRNA)的脱离使得大亚基结合到小亚基上。与原核细胞的情况相同, 大亚基的结合刺激了eIF5B-GTP(与原核的IF2相似)的水解, 导致剩余的起始因子释放。这些事件的结果是, Met-tRNAiMet被置于新的80S起始复合体(80S initiation complex)的P位点。
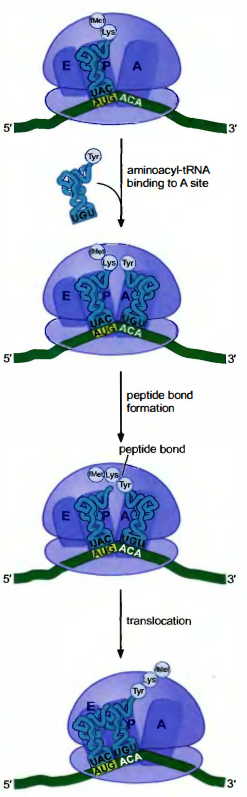
[!CAUTION] 真核翻译起始和原核不同之处!!!(补充)
- 起始密码子和起始氨酰tRNA不同
原核生物的起始密码子有AUG、GUG、UUG, 起始氨酰tRNA为fMet-tRNAifMet, 真核生物起始密码子为AUG, 起始氨酰tRNA为Met-tRNAMet。- 原核生物mRNA上有能与17SrRNA配对的SD序列, 而真核生物没有这样的序列。
- 起始识别机制不同: 原核生物是由小亚基与mRNA的SD序列, 起始因子帮助mRNA的起始密码子与AUG定位于小亚基的P位点。真核生物核糖体小亚基被募集到mRNA的5, 帽子, 一旦结合到mRNA上, 核糖体小亚基沿5, -3, 方向移动直至扫描到第一个AUG序列, 该密码子被识别为起始密码子。
- 起始密复合体的形成机制不同。
- 原核生物起始因子有4个, 真核生物中有十几个。
- 原核生物起始过程中不需要消耗ATP解开二级结构, 而真核生物需要消耗ATP。
翻译延伸
为了使氨基酸能正确加入, 有3个关键的事件是必须发生的:
- 在A位点上的密码子的指导下, 正确的氨基酰tRNA置于A位点上;
- A位点的氨基酰tRNA与P位点的肽酰tRNA上的肽链形成肽键, 这一肽转移酶反应导致多肽链从P位点的tRNA上转移到A位点的负载tRNA的氨基酸残基上;
- 形成的A位点的肽酰tRNA和相应的密码子必须易位至P位点, 以使核糖体为下一循环的密码子识别和肽键形成做好准备。
氨基酰tRNA由延伸因子EF-Tu送达A位点
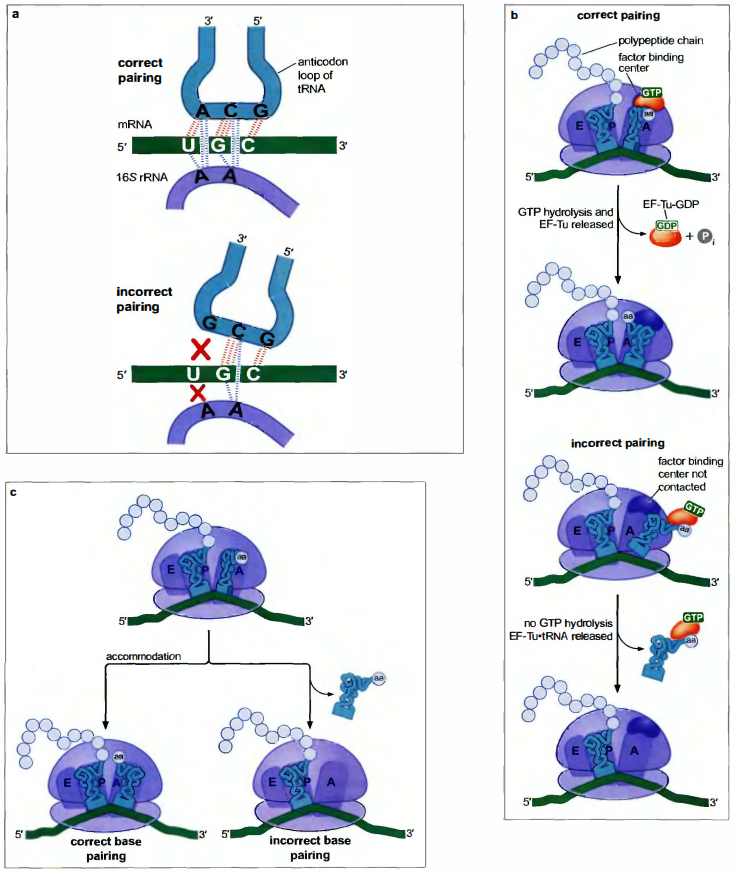
只有当EF-Tu与GTP结合后, EF-Tu才能结合氨基酰tRNA。结合氨基酰tRNA的EF-Tu不能有效地水解GTP。激活EF-Tu GTP水解酶活性的”扳机”是与大亚基加入起始复合体时激活IF2 GTP水解酶的大亚基的同一个结构域。这一结构域称为因子结合中心(factor binding center)。只有当tRNA置于A位点和正确的密码子-反密码子配对后, EF-Tu才能与因子结合中心相互作用。然后, EF-Tu水解结合的GTP, 并从核糖体中释放出来。
核糖体利用多种选择机制排斥错误配对的氨基酰tRNA
选择正确的氨酰tRNA的根本原则在于负载tRNA和核糖体A位点的密码子之间的碱基配对。
至少有3中不同的机制促成了翻译的高准确率:
- 其中一个关于密码子识别忠实性的机制涉及小亚基16SrRNA的两个相连腺嘌呤残基。这两个腺嘌呤与反密码子和密码子的前两个碱基之间每个正确配对所形成的小沟形成紧密的相互作用。相邻的16SrRNA的A残基不能识别G: C或A: U配对, 因而认为任何一对都是正确的。相反, 非Watson-Crick碱基配对所形成的小沟就不能被(16SrRNA的)这两个腺嘌呤识别, 导致对非正确tRNA的亲和力明显降低。这些作用的净结果是, 正确配对的tRNA从核糖体解离的速度要远低于非正确配对的tRNA。
- 第二个有助于保证正确的反密码子-密码子配对的机制涉及EF-Tu的GTP酶活性。EF-Tu从tRNA的释放需要GTP的水解, 这一作用对正确的反密码子-密码子配对是非常敏感的。即使只有一个碱基的不配对也将导致EF-Tu的GTP酶活性的急剧下降。
- 第三个保证碱基配对正确性的机制是EF-Tu释放后的一个校正机制。当负载tRNA与EF-Tu-GTP的复合体进入A位点时, 它的3’端远离肽键形成的位点。为了成功地进行肽转移酶反应, 氨基酰tRNA必须旋转进入到大亚基的肽转移酶中心, 这一过程称为入位(accommodation)。非正确配对的tRNA在入位过程中经常从核糖体上脱离下来。有假设认为氨基酰tRNA的旋转为密码子和反密码子的作用带来了张力, 而只有正确配对的反密码子才能维持这种张力。
核糖体是一种核酶
这一反应由RNA, 尤其是大亚基的23S rRNA组分来催化。23S rRNA与处于A和P位点的tRNA的CCA末端之间的碱基配对, 帮助氨基酰tRNA的α氨基基团攻击结合于肽酰-tRNA的多肽的碳基团。
肽转移酶中心的核苷酸接受了氨基酰tRNA的α氨基基团的一个氢原子, 使得相连的氮原子变成了极强的亲核基团。
肽键形成启动大亚基中的易位反应
易位(translocation): 一旦肽转移酶反应开始发生, P位点的tRNA就被脱去氨基(不再结合氨基酸), 而多肽链则连接到A位点的tRNA上。要使肽链延伸的新一轮循环得以发生, P位点的tRNA必须移至E位点, 而A位点的tRNA必须移至P位点。同时, mRNA也必须移动3个核苷酸, 以使下一个密码子暴露出来。这些移动是在核糖体内协调进行的, 统称为易位。
易位的起始步骤是与肽转移酶反应偶联的。一旦多肽链移至A位点的tRNA, 这个tRNA的3’端就移到大亚基的P位点上。相反, tRNA的反密码子部分仍然留在A位点内。相似地, 这时已脱氨基的P位点的tRNA位于大亚基的E位点和小亚基的P位点上。
EF-G通过稳定中间体的方法推动易位
易位的完成需要第二个称为EF-G的延伸因子的作用。当EF-G-GTP结合后, 它与大亚基的因子结合中心相作用, 刺激GTP的水解。
GTP的水解改变了EF-G-GDP的构象, 允许它进入小亚基并刺激A位点tRNA的易位。当易位完成后, 核糖体构象的改变极大地降低了其对EF-G-GDP的亲和力, 允许这一延伸因子从核糖体中释放出来。这些事件一起导致A位点的tRNA易位至P位点, P位点的tRNA易位至E位点, 以及mRNA正好移动3个核苷酸。
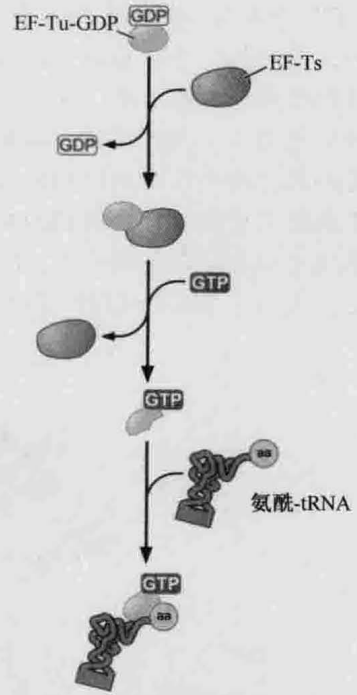
EF-Tu-GDP和EF-G-GDP在参加新一轮延伸之前必须将GDP换成GTP
对EF-G来说, 这是一个简单的过程, 因为GDP与EF-G的亲和力要低于GTP。因此GTP水解后, GDP和磷酸释放出来, 游离的EF-G很快与一个新的GTP分子相结合。而对EF-Tu而言, 需要另一个蛋白质来完成GDP到GTP的交换。延伸因子EF-Ts是EF-Tu的GTP交换因子(GTP exchange factor)。EF-Tu-GDP从核糖体释放出来后, EF-TS与EF-Tu结合, 促使GDP脱落。随后, GTP结合EF-Tu-EF-Ts复合体, 导致它分解为游离的EF-Ts和EF-Tu-GTP。最后, EF-Tu-GTP结合一个负载的tRNA,重新形成EF-Tu-GTP-氨酰-tRNA复合体, 再一次准备将新的负载tRNA送至核糖体。
形成肽键的一个循环要消耗两个GTP分子和一个ATP分子
一个ATP分子被氨酰-tRNA合成酶消耗, 形成了连接氨基酸和tRNA的高能酰基键。这一高能酰基键断裂(所释放的能量)驱动了产生肽键的肽转移酶反应。
第二个三磷酸核苷酸(GTP)分子在EF-Tu将负载tRNA运送至核糖体的A位点和确保正确的密码子-反密码子的识别过程中被消耗。最后。
第三个三磷酸核苷酸(GTP)在EF-G介导的易位过程中被消耗。
这样, 一个肽键的形成就消耗了细胞两个GTP分子和一个ATP分子, 翻译延伸过程的每一步消耗一个三磷酸核苷酸。这三个分子中, 只有一个(ATP)与肽键的形成有关, 另外两个(GTP)分子的能量用于保证翻译的精确有序。
翻译终止
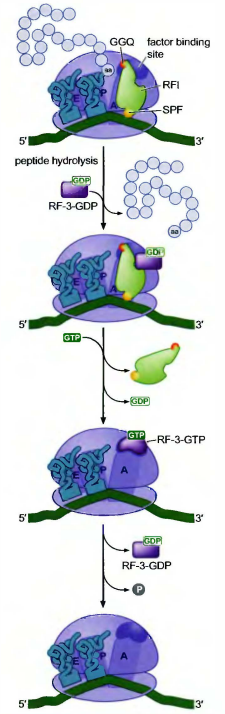
释放因子在终止密码子的作用下终止翻译
释放因子(release factor, RF): 识别终止密码子的一种蛋白质因子, 这些因子激活多肽链从肽酰tRNA中释放的水解反应。
有两类释放因子:
- I类释放因子识别终止密码子, 并催化多肽链从P位点的tRNA中水解释放出来。
原核细胞有两种I类释放因子: RF1和RF2。RF1识别终止密码子UAG, RF2识别终止密码子UGA, 两者皆可识别第三个终止密码子UAA。真核细胞只有一种能识别3个终止密码子的Ⅰ类释放因子: eRF1。 - II类释放因子在多肽链释放后刺激Ⅰ类因子从核糖体中解离开来。原核细胞和真核细胞都只有一种Ⅱ类因子, 分别是RF3和eRF3。与EF-G, EF-Tu和其他翻译因子一样, II类释放因子也是由GTP调节的。
I类释放因子的一小段区域识别终止密码子并催化多肽链的释放
释放因子中的识别区域: 3个氨基酸负责识别终止密码子的特异性。这3个氨基酸组成的区域代表着肽反密码子。
所有的I类因子共有一段保守的三氨基酸(GGQ)序列, 这一序列对多肽链的释放是关键的。
GDP/GTP交换和GTP水解调控II类释放因子的功能
在I类RF刺激多肽释放后, 核糖体和I类因子的构象改变诱导RF3交换GDP而结合GTP。RF3与GTP的结合导致与核糖体之间的高亲和力相互租用的形成, 替代了I类分子与核糖体的结合, 这一变化还使得RF3与大亚基的因子结合中心结合。与参与翻译的其他GTP结合蛋白一样, 这个作用刺激GTP的水解。在(与核糖体结合的)Ⅰ类因子不存在的情况下, RF3-GDP与核糖体的亲和力较弱而很快从核糖体中释放出来
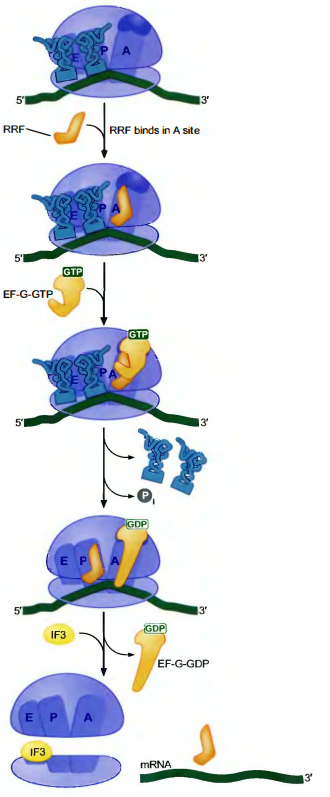
核糖体循环因子模仿tRNA
核糖体循环(ribosome recycling): 在多肽链和释放因子离开核糖体后, 核糖体仍然结合mRNA,并留有两个脱氨基的tRNA(在P位点和E位点)。为了参与新一轮的多肽合成, tRNA和mRNA必须离开核糖体而核糖体也必须分解为大、小亚基。这几个事件统称为核糖体循环。
在原核细胞中, 一种称为核糖体循环因子(ribosome recycling factor, RRF)在多肽释放后与EF-G和IF3共同作用来推动核糖体循环。RRF与空位的A位点结合, 并模仿tRNA。RRF也募集EF-G-GTP到核糖体上, 在那些模仿延伸过程中EF-G功能的事件中, EF-G引发结合在P位点和E位点的空载tRNA的释放。一旦tRNA从核糖体中脱离, EF-G-GDP和RRF同mRNA一起也从核糖体中释放出来。IF3(起始因子)也可能参与了mRNA的释放, 而且它对核糖体分解为大、小亚基是必需的。最后结果形成个与IF3(而非tRNA或mRNA)结合的小亚基和一个游离的大亚基。这些游离的核糖体就可以参与新一轮的翻译。
翻译的调控
依赖于翻译过程的mRNA和蛋白质稳定性调节
mRNA会以一定的频率出现突变和损伤。这样的缺陷mRNA可能由转录错误造成, 也可能由其合成后的损伤造成。这样的缺陷mRNA可能产生不完整和不正确的蛋白质, 它们可能给细胞带来负面作用。翻译过程可以识别缺陷的mRNA,并予以去除或将其蛋白质产物去除。
SsrA RNA援救翻译着断裂mRNA的核糖体(原核细胞)
对于在可读框中缺失了终止密码子的mRNA上进行翻译的核糖体将被怎么处理呢?
如果没有一定的机制使核糖体脱离这些缺陷mRNA,那么很多核糖体将永久地受困, 不能参与多肽的合成。
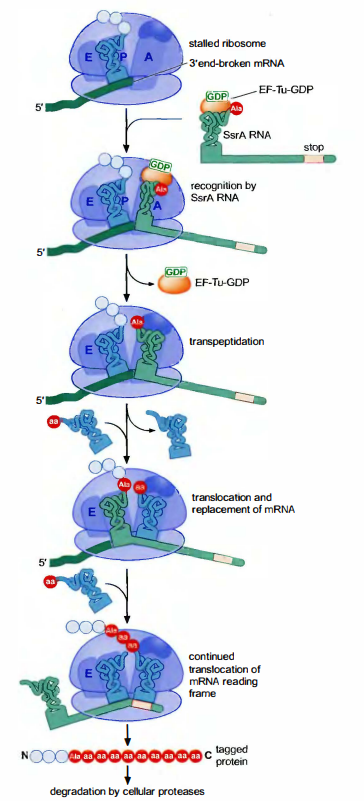
在原核细胞中, 由一种嵌合的RNA分子解救这样的核糖体, 这种RNA分子部分是tRNA、部分是mRNA,称为tmRNA。
SsrA RNA是一个由457个核苷酸组成的tmRNA,其3’端的区域与tRNA Ala非常相似。这一相似性使得SsrARNA可以负载Ala并结合EF-Tu-GTP。当一个核糖体停滞于mRNA的3’端时, SsrAAla-EF-Tu-GTP结合于核糖体的A位点并参与肽转移酶反应, 就如其他的tRNA一样。肽酰-SsrA RNA的易位导致缺陷的mRNA从核糖体中释放出来。SsrA RNA的易位也导致这一RNA的一部分进人核糖体的mRNA结合隧道。SsrA RNA结合的最终结果是, 当缺陷mRNA从核糖体中释放出来时, 不完整多肽的碳端融合了一个10个氨基酸的”标签”, 而核糖体重新进人翻译的循环过程中。这种10个氨基酸的”标签”被细胞的蛋白水解酶识别, 蛋白水解酶很快地将这一标签以及它附着的多肽降解。这样, 缺陷mRNA的多肽产物很快就被清除, 从而防止了其对细胞可能带来的伤害。
SsrA RNA为什么只能与停滞的核糖体结合呢?
SsrA RNA的体积巨大(它比标准的tRNA大4倍以上), 在正常的延伸过程中它不能结合于A位点。相反, 当mRNA的3’端缺失时, A位点就有更大的体积来容纳较大的RNA。因此, 只有核糖体停滞于mRNA的3’端, 才能为SsrARNA提供潜在的结合位点。
真核细胞降解不完整的或终止密码子提前的mRNA
真核细胞降解不完整的或终止密码子提前的mRNA有三种机制:
- 无义密码子介导的mRNA衰减(nonsense mediated mRNA decay):
当一个mRNA分子含有提前的终止密码子时, 这一mRNA很快被降解, 此过程称为无义密码子介导的mRNA衰减。
在哺乳动物细胞中, 对含有提前的终止密码子的mRNA的识别依赖于聚集在mRNA可读框内的蛋白复合体。这些外显子拼接复合体是剪接过程所产生的产物并聚合在mRNA上的, 并正好坐落于每一外显子外显子结合处的上游。
当第一个核糖体翻译某个mRNA时, 这些复合体在mRNA进人核糖体的解码中心时被置换出来。但是, 如果mRNA存在一个提前的终止密码子(源于基因的突变或转录与剪接过程中的错误), 那么核糖体就在这些复合体被置换之前从mRNA上脱离出来。
在这些情况下, 外显子拼接复合体和eRF3将结合于提前终止的核糖体并募集一系列的蛋白。这些蛋白募集并且/或者激活多种酶对mRNA进行剪切, 并去除mRNA 5’端帽结构, 或3’端的poly-A尾结构。由于mRNA通常就是受5”端帽结构保护来防止降解的, 因而这些过程令未受保护的5’或3’端暴露出来, 从而导致mRNA很快地被5’→3’和3’→5’核酸外切酶所降解。
- 无终止密码子介导的衰减(nonstop mediated decay):
另一种挽救正在翻译缺乏终止密码子的mRNA的核糖体的机制, 称为无终止密码子介导的衰减。
与原核细胞不同, 真核细胞的mRNA以多聚A尾结束。当缺失终止密码子的mRNA翻译时, 核糖体就会翻译多聚A尾(因为没有终止密码子在多聚A尾之前终止翻译)。这就导致了蛋白质的末端添加了多个赖氨酸(AAA是赖氨酸的密码子),并使核糖体停止在mRNA的3’端。
停止的核糖体与eRF1和eRF3(Dom34和Hbs1l)蛋白结合, 从而促使核糖体的解离以及释放肽酰-tRNA和mRNA。第二类eRF3相关因子, Ski7蛋白, 能够募集3’→5’核酸外切酶降解”无终止密码子”的mRNA。
无终止密码子介导的衰减过程中, 没有终止密码子的mRNA也可以被核酸内切酶切断。在羰基末端含有多聚赖氨酸的蛋白质是不稳定的, 导致无终止密码子的mRNA表达的蛋白质被快速降解。
- 末端终止介导的衰减(no-go decay): 与无终止密码子介导的衰减类似, 它被称为末端终止介导的衰减。
这种机制能够识别在一条mRNA上停滞的核糖体。这种情况会发生在一个mRNA的编码区上出现了稳定的二级结构的时候, 或者一连串的密码子对应的tRNA在某个细胞中不够用的时候。
无义密码子介导, 无终止密码子介导和末端终止介导的mRNA衰减都有一个引人注目的特性:(三个机制都依赖翻译过程)都需要通过受损mRNA的翻译过程去检测出缺陷的mRNA并对其降解。在没有进行翻译过程的情况下, 受损的mRNA并不被很快地降解, 而且具有正常的稳定性。因此三个机制并不是直接作用, 真核细胞需要依赖翻译的机制来校正它们的mRNA。
补充
蛋白质前体的加工
新生的多肽链大多数是没有功能的, 必须经过加工修饰才能转变为有活性的蛋白质。
- N端fMet或Met的切除
细菌蛋白质氨基端的甲酰基能被脱甲酰化酶水解,不管是原核生物还是真核生物,N端的甲硫氨酸往往在多肽链合成完毕之前就被切除。有些动物病毒如脊髓灰质炎病毒的mRNA可翻译成很长的多肽链, 含多种病毒蛋白,经蛋白酶在特定位置上水解后得到几个有功能的蛋白质分子。 - 二硫键的形成
mRNA中没有胱氨酸的密码子, 而不少蛋白质都含有二硫键, 这是蛋白质合成后通过两个半胱氨酸的氧化作用生成的。二硫键的正确形成对稳定蛋白的天然构象具有重要的作用。 - 特定氨基酸的修饰
氨基酸侧链的修饰作用包括磷酸化(如核糖体蛋白)、糖基化(如各种糖蛋白)、甲基化(如组蛋白、肌肉蛋白质),乙酰化(如组蛋白), 泛素化(多种蛋白), 羟基化(如胶原蛋白)和羧基化等。组蛋白N端35个残基中可能出现包括磷酸化、甲基化、乙酰化和泛素化在内的多种修饰。糖蛋白主要是通过蛋白质侧链上的天冬氨酸、丝氨酸、苏氨酸残基加上糖基形成的,胶原蛋白上的脯氨酸和赖氨酸多数是羟基化的。实验证明,内质网可能是蛋白质N-糖基化的主要场所。
(1)磷酸化(phosphorylation): 主要由多种蛋白激酶催化,发生在丝氨酸、苏氨酸和酪氨酸等3种氨基酸的侧链。
(2)糖基化(gycosylation): 是真核细胞蛋白质的特征之一。大多数糖基化是由内质网中的糖基化酶(glycosylase)催化进行的。所有的分泌蛋白和膜蛋白几乎都是糖基化蛋白质。
(3)甲基化(methylation): 蛋白质的甲基化是由N-甲基转移酶催化的, 该酶主要存在于细胞质基质内。甲基化包括发生在Arg、His和Gln的侧基的N-甲基化以及Glu和Asp侧基的0-甲基化。(4)乙酰化(acetylation)N- 乙酰转移酶催化多肽链的N端乙酰化。发生在赖氨酸侧链上的ε-NH2。4. 切除新生肽链中的非功能片段
新合成的胰岛素前体是前胰岛素原,必须先切去信号肽变成胰岛素原,再切去B-肽,才变成有活性的胰岛素。不少多肽类激素和酶的前体都要经过加工才能变为活性分子, 如血纤维蛋白原、胰蛋白酶原经过加工切去部分肽段才能成为有活性的血纤维蛋白、胰蛋白酶。蜂毒素能溶解动物细胞,也能溶解蜜蜂自身的细胞, 所以只能在细胞内合成没有活性的前毒素,分泌进人刺吸器后,其N端的22个氨基酸残基被蛋白酶水解, 生成有毒性的功能蛋白质。一般说来,由多个肽链及其他辅助成分构成的蛋白质,在多肽链合成后还需经过多肽链之间以及多肽链与辅基之间的聚合过程,才能成为有活性的蛋白质。
考点补充
名词补充