分子生物学 第十三章 转录机制
1
2
3
4
5
6
7
8
9 ______ __ __
/\__ _\ __ /\ \__ __ /'\_/`\ /\ \ __
\/_/\ \/ _ __ __ ___ ____ ___ _ __ /\_\ _____\ \ ,_\/\_\ ___ ___ /\ \ __ ___\ \ \___ __ ___ /\_\ ____ ___ ___
\ \ \/\`'__\/'__`\ /' _ `\ /',__\ /'___\/\`'__\/\ \/\ '__`\ \ \/\/\ \ / __`\ /' _ `\ \ \ \__\ \ /'__`\ /'___\ \ _ `\ /'__`\ /' _ `\/\ \ /',__\ /' __` __`\
\ \ \ \ \//\ \L\.\_/\ \/\ \/\__, `\/\ \__/\ \ \/ \ \ \ \ \L\ \ \ \_\ \ \/\ \L\ \/\ \/\ \ \ \ \_/\ \/\ __//\ \__/\ \ \ \ \/\ \L\.\_/\ \/\ \ \ \/\__, `\/\ \/\ \/\ \
\ \_\ \_\\ \__/.\_\ \_\ \_\/\____/\ \____\\ \_\ \ \_\ \ ,__/\ \__\\ \_\ \____/\ \_\ \_\ \ \_\\ \_\ \____\ \____\\ \_\ \_\ \__/.\_\ \_\ \_\ \_\/\____/\ \_\ \_\ \_\
\/_/\/_/ \/__/\/_/\/_/\/_/\/___/ \/____/ \/_/ \/_/\ \ \/ \/__/ \/_/\/___/ \/_/\/_/ \/_/ \/_/\/____/\/____/ \/_/\/_/\/__/\/_/\/_/\/_/\/_/\/___/ \/_/\/_/\/_/
\ \_\
\/_/
RNA聚合酶和转录周期
真核和原核RNA聚合酶的区别
RNA聚合酶具有不同形式, 但有许多共同特性
| 真核生物 | 原核生物 | |
|---|---|---|
| 结构 | 真核生物RNApol有I、II、III三种,各由多个亚基组成 | 原核生物RNA-pol是由多个亚基构成的。a2ββ’称为核心酶,a2ββ’o称为全酶 |
| 功能 | 专一性地转录不同的基因,产生不同的产物,分别转录rRNA,hnRNA和小型分子的RNA(5s-rRNA, tRNA和snRNA) | 转录几乎所有蛋白质编码基因的酶 |
| RNA聚合酶 | 细胞内定位 | 转录产物 |
|---|---|---|
| RNA pol I | 核仁 | rRNA(18S rRNA、5.8S rRNA、28S rRNA) |
| RNA pol II | 核质 | hnRNA、snRNA |
| RNA pol III | 核质 | tRNA、U6 snRNA、5S rRNA |
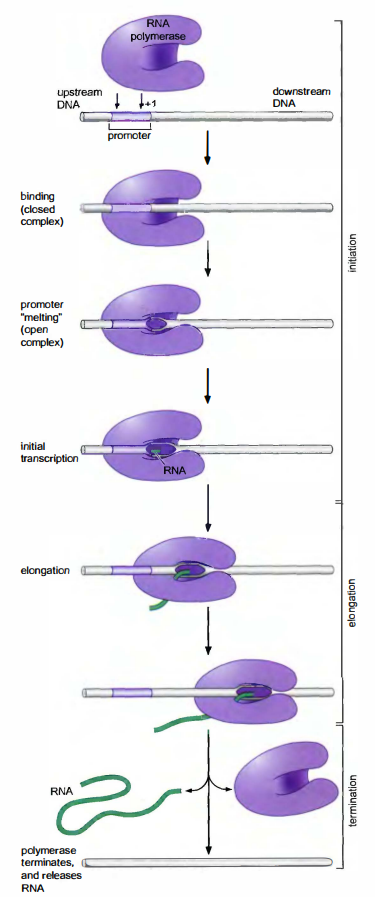
由RNA聚合酶进行的转录是由一系列步骤完成的
- 起始(initiation):启动子(promoter):是最初结合 RNA 聚合酶的DNA 序列(在很多情况下是与转录起始因子一起结合的)。启动子-聚合酶复合体一旦形成就发生结构改变,使起始过程继续进行。
转录起始包括三个定义明确的步骤:- 第一步是聚合酶与启动子初步结合形成所谓的闭合式复合体(closed complex),这时DNA还保持着双链状态,聚合酶结合在螺旋的一个面上。
- 第二步是闭合式复合体经过转变而成为开放式复合体(open complex),DNA 双链在起始位点周围大约 14bp 的距离内分开以形成转录泡。
- 第三步,最初的大约 10 个左右的核糖核苷酸的结合是一个效率相当低的过程,在这一阶段,聚合酶经常释放出很短的转录物(每一个都短于 10 个左右的核苷酸),然后再重新开始合成。
启动子逃离:一旦某个聚合酶超过了 10bp,就可以说它逃离了启动子。这时,它已经形成了一个稳定的三重复合体(stable ternary complex),包括酶、DNA 和 RNA,完成了向延伸的转变。
- 延伸(elongation):一旦 RNA 聚合酶已经合成了一小段 RNA(10 个碱基左右),转录便进入了延伸阶段。这一转变需要聚合酶构象的进一步改变,以使其能够更牢固地与模板结合。在延伸阶段,RNA聚合酶除催化 RNA 的合成外,还执行着多项重要任务。它解开前方的 DNA,又使后方的DNA 重新复性;在移动的同时,逐步分开正在延长的 RNA 链与模板的配对;而且还执行校正的功能。
- 终止(termination):一旦 RNA 聚合酶转录了整个基因(或整组基因),它必须停下来并释放RNA产物。这一步骤称为终止。在某些细胞中,存在着特定的、已研究清楚的引发终止的序列;而在其他一些细胞中,是什么使聚合酶停止转录并从模板上分离,还不十分清楚。
细菌的转录周期
细菌的启动子在强度和序列上是多样的,但是具有某些明确的特征
RNA聚合酶的全酶(holoenzyme): 一个称为σ的起始因子的添加, 使得核心酶转变为只在启动子处起始转录的形式。该酶的这种形式称为RNA聚合酶的全酶。
在大肠杆菌中, 最常见的σ因子称为σ70。含有σ70的聚合酶识别的启动子具有以下共同的特征性结构: 两段6个核苷酸长的保守序列, 被长为17~19个核苷酸的非特异序列所分割。这两段序列称为-35和-10区域(region)或元件(element)。
共有序列(consensus sequence): 指多种原核基因启动序列特定区域内, 通常在转录起始点上游-10及-35区域存在一些相似序列。具有与共有序列更近似的序列的启动子通常要比那些匹配较差的启动子”更强”。
启动子的强度: 是指一个启动子在一定的时间内可以起始多少个转录物, 主要受以下因素影响:
- 启动子最初与聚合酶的结合程度;
- 对异构化作用的支持效率;
- 此后聚合酶逃离的难易程度。
UP元件(UP-element): 在一些较强的启动子中, 如在指导核糖体RNA(rRNA)基因表达的启动子中, 发现了一个额外的与RNA聚合酶结合的DNA元件, 称为UP元件, 它可以通过提供额外的特异性相互作用而增强聚合酶与DNA的结合。
另一类型的σ70启动子缺乏-35区域, 而是由一个所谓的”延长的-10区域”元件。与标准的-10区域相比, 这一元件在其上游末端有个额外的短序列元件。聚合酶与这一额外序列元件之间的联系, 补偿了-35区域的缺失。
鉴别子(discriminator): 在紧挨着-10元件的下游发现了一个新的结合RNA聚合酶的DNA元件。这个新元件称为鉴别子。鉴别子与聚合酶之间相互作用的强度影响酶与启动子复合体的稳定性, 从而影响转录效率。
σ因子介导聚合酶与启动子的结合
σ70因子可以分为4个区域, 称为σ区域1~σ区域4。
区域4内的两个螺旋形成一个常见的DNA结合基序, 称为螺旋-转角-螺旋。其中一个螺旋插入到-35区域的大沟中并与该区的碱基相互作用, 另一个则从大沟的顶部横过, 与DNA骨架接触。
-10区域也被一个α螺旋所识别。但是其相互作用尚未彻底研究清楚而且更为复杂。
延长的-10元件, 如果存在的话, 被σ区域3中的一个α螺旋所识别, 此螺旋与组成这一元件的两个特异性的碱基对接触。
鉴别子由σ区域1、2识别。
与启动子内的其他元件不同, UP元件并不被σ因子所识别, 而是由α亚基的羧基末端域(αCTD)来识别。
σ因子和α亚基将RNA聚合酶核心酶招募到启动子上。
向开放式复合体的转变过程涉及RNA聚合酶和启动子DNA的结构变化
启动子DNA的变化: DNA双链的打开, 发生在-11和+3之间的区域。
异构化作用(isomerization): 从闭合式复合体到开放式复合体的转变过程涉及聚合酶的结构变化, 以及DNA双链的打开, 从而暴露出模板链和非模板链。对于含有σ70因子的细菌聚合酶, 这一转变常被称为异构化作用。异构化是DNA-聚合酶复合物构象自发地变成一种在能量上更具有优势的形式, 无需ATP水解提供能量。
聚合酶发生的结构变化:
- 位于聚合酶前部的钳子牢固地钳压在下游dna上;
- σ的n端区域(区域1.1)的位置有一个重要的移动, 在未结合dna时, σ区域1.1处于全酶的活性中心裂隙内部, 阻断着开放式复合体中模板dna链经过的路线。在开放式复合体中, 区域1.1移动了大约50ẳ的距离, 出现在酶的外部, 使得dna可以进到裂隙中。
转录由RNA聚合酶起始, 不需要引物
起始要求: RNA聚合酶可以起始新RNA链的合成, 不需要引物
- 起始核苷酸(通常是一个A)被带到活性位点;
- 聚合酶必须与一条或者多条DNA模板链、起始核苷酸以及第二个核苷酸建立特异性的相互作用, 严格控制其保持正确的方向, 使其可以对引入的核苷三磷酸进行化学反应。
RNA聚合酶在进入延伸阶段之前会先合成几段短的RNA
流产性起始(abortive initiation): 一旦核苷酸进入了活性中心裂隙并且RNA开始合成, 聚合酶会合成一些长度小于10个核苷酸的RNA分子, 为了使合成长度超过大约10个核苷酸的RNA链, σ的这一区域必须从此位置上被逐出, 这个过程需要聚合酶尝试几次才能成功, 称为流产性起始。
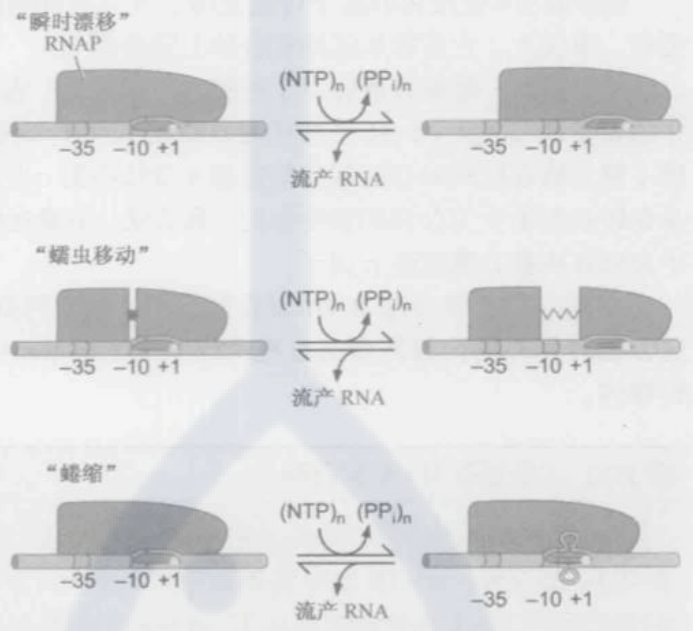
转录起始阶段, RNA聚合酶保持位置不变并将下游DNA拉向自己
- “瞬时漂移”(transient excursion)模型: 提出RNA聚合酶正向反向的瞬时迁移循环。聚合酶被认为离开启动子沿着DNA模板移动一小段, 合成一小段转录物, 然后流产释放转录物, 再回到其在启动子的原始位置。
- “蠕虫移动”(inchworming)模型: 认为在聚合酶内有一个具伸缩性的元件, 使得酶前方含有活性位点的部件向下游移动, 合成一小段转录物, 然后流产, 再收缩回到其仍留在启动子上的酶主体。
- “蜷缩”(scrunching)模型: 认为位于固定位置的、与启动子结合的、聚合酶下游的DNA被酶拉进来。于是在酶的内部堆积的DNA以单链”泡”的形式逗留。
延伸聚合酶是一个边合成边校对RNA的向前行进的机器
RNA聚合酶的功能:
- DNA通过延伸酶的形式与通过开放式复合体相似, 双链DNA在两个钳子之间进入酶的前部。在催化裂隙的开口处, 两条链分别沿着不同的路径穿过酶, 然后从各自的通道离开酶, 并在延伸聚合酶(elongationpolymerase)的后面重新形成双螺旋结构。核苷酸通过其固定的通道进入活性位点, 并在模板DNA链的引导下添加到增长中的RNA链上。
- 除了所有这些功能外, RNA聚合酶还执行着两项校对(proofreading)功能:
- 焦磷酸化编辑(pyrophosphorolytic editing): 聚合酶利用它的活性位点, 在一个简单的逆向反应中, 通过加入PPi, 催化错误插入的核糖核苷酸的去除。
- 水解编辑(hydrolytic editing): 聚合酶倒退一个或更多的核苷酸并切开RNA产物, 去除掉错误的序列。水解编辑是由一些Gre因子激发的。Gre因子能够增强水解编辑的功能, 同时也作为延伸刺激因子, 它们确保聚合酶能够有效地延伸并帮助克服转录在较难转录的序列处”停滞不前”。
RNA聚合酶可能变得停滞不前, 需要挪走
RNA聚合酶停止的原因: 停滞不前的一个常见理由是一条损坏的DNA链。如果被转录的基因是必不可少的, 停滞不前的聚合酶不能制造有效的产物, 并且对试图转录同一个基因的其他聚合酶造成障碍。
解决方法:
TRCF: 具有ATPase活性,它结合聚合酶上游的双链DNA,并且利用ATPase传动器(motor)沿着DNA移动, 直到遇到停止的RNA聚合酶。碰撞把聚合酶往前推, 或是使它重新开始延伸, 更常见的是, 使RNA聚合酶、模板DNA、和RNA转录物的三重复合体解离。这样就结束了酶的转录, 但修复酶和其他RNA聚合酶开辟了道路。
转录由RNA序列内部信号终止
转录终止有两种方式:
- 异常终止: 由损坏的DNA或其他不曾预料到的障碍引起, 通过TRCF的作用解决;
- 正常终止: 在基因末端, 转录终止是一个正常且重要的过程, 由终止子引发。
- 终止子(terminator): 引发延伸聚合酶从DNA上脱离并且释放出它已经合成的RNA链的序列。在细菌中, 终止子有两种类型: Rho-非依赖型和Rho-依赖型。第一种类型不需要其他因子的参与就可以引发聚合酶的终止反应, 第二种类型, 需要一个称为Rho的额外的蛋白质来诱发终止反应。
- Rho依赖型终止(Rho-dependent): Rho依赖型终止有一些定义不太明确的RNA元件, 被称为rut位点。需要Rho蛋白诱发终止, Rho是一个具有6个相同亚基的环状蛋白质, 在单链RNA离开聚合酶时与之结合。此蛋白质还具有ATPase活性, 一旦与转录物结合, 它就利用ATP水解产生的能量来诱发终止。
- Rho引导到特定的RNA分子上: 首先, Rho结合的位点有某种特异性。特异性的第二层次是Rho无法与任何正在被翻译的转录物结合。
- Rho-非依赖型终止子(Rho-independent): 也称为固有终止子(intrinsic terminator), 由两个序列元件组成: 一段短的反向重复序列(大约20个核苷酸), 其后是一段大约8个A: T碱基对的序列。转录后才会影响聚合酶, 也就是说它们是以RNA而不是DNA起作用的, 合成的RNA能够通过自身碱基配对形成一个茎环结构(stem-loop)。发夹的形成破坏了延伸复合体, 从而引起终止。
- 发夹或是把聚合酶往相对于DNA和RNA的方向推, 或是把转录物从聚合酶上硬扯下来, 或是诱发聚合酶的构象改变而导致终止。
真核生物的转录
真核生物有三个不同的聚合酶(Pol I、Pol II和Pol III), 而细菌只有一个。还有, 细菌只需要一个额外的起始因子(σ), 而真核生物中有效的和启动子特异的起始则需要几个起始因子。这些起始因子称为通用转录因子(general transcription factor, GTF)。
通用转录因子(general transcription factor, GTF): 真核生物中有效的、启动子特异的转录起始所需的因子。在体外, 通用转录因子和RNA聚合酶II构成, 从一个DNA模板(不含组蛋白)上起始转录所需的全部蛋白。
在体内, 真核细胞内的DNA模板是整合在核小体内的。在这种环境下, 通用转录因子就无法启动有意义的表达, 而是需要额外的因子参与, 包括所谓的”中介蛋白复合体”(mediator complex)和DNA结合调节蛋白, 并常常包括染色质修饰酶。
RNA聚合酶II的核心启动子由4个不同序列的元件组合而成
核心启动子(core promoter): 是指在体外检测时, 真核细胞PolⅡ机器精确地起始转录所需要的最少的一组序列元件。在PolⅡ核心启动子中发现的4个元件分别是TFⅡB识别元件(TFⅡBrecognitionelement, BRE)、TATA元件(或盒子)、起始密码子(initiator, Inr)和下游启动子元件(downstreampromoterelement, DPE)。
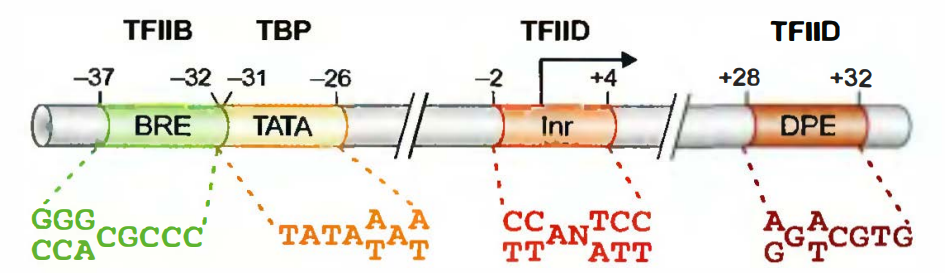
在核心启动子之外(而且通常是在其上游)存在一些在体内进行有效转录所需的其他序列元件。这些元件包括: 启动子最近元件(promoter proximal element), 上游激活物序列(upstream activatorsequence, UAS), 增强子(enhancer), 以及一系列的称为沉默子(silencer)、边界元件(boundary element)和绝缘子(insulator)的抑制元件。
调节序列(regulatory sequence): 在核心启动子之外的(而且通常是在其上游), 在体内进行有效转录所需的其他序列元件。
RNA聚合酶Ⅱ在启动子上和通用转录因子形成前起始复合体
前起始复合体(pre-initiation complex): 一起结合在启动子上准备开始转录的一套完整的通用转录因子和聚合酶称为前起始复合体。形成的位置: PolII启动子包含一个TATA元件(大约在转录起始位点上游30个碱基对处)。
TATA元件是由叫做TFIID的通用转录因子所识别的, TFIID实际上是一个多亚基复合体。TFIID中与TATA序列结合的成分称为TBP(TATA binding protein)。此复合体中的其他亚基称为TAF,即TBP关联因子(TBP-associated factor)。某些TAF识别其他启动子核心元件, 如Inr、DPE和DCE, TFIID是启动子识别和建立前起始复合体必不可少的因子。
前起始复合体的组装:TBP一旦结合到DNA上, 就使TATA序列极大地扭曲变形。形成的TBP-DNA复合体提供了一个平台, 把其他通用转录因子和聚合酶本身募集到启动子上。在体外, 这些蛋白质按照下列顺序在启动子处组装: TFIIA、TFIIB、TFIIF与聚合酶一起, 然后是TFIIE和TFIIH。包含这些成分的前起始复合体形成后, 启动子就开始解旋。真核细胞中的启动子解旋需要ATP的水解并由TFIIH介导, 是该因子的类解旋酶活性刺激了启动子DNA的解开。
启动子逃离需要聚合酶“尾巴”的磷酸化
真核细胞启动子逃离两个步骤(细菌没有): 一个是ATP水解, 另一个是聚合酶的磷酸化: PolII的大亚基有一个C端域(CTD), 延伸成一个”尾巴”。CTD包含一连串重复的七肽序列: Tyr-Ser-Pro-Thr-Ser-Pro-Ser。每一个重复序列都包含特定蛋白激酶的磷酸化位点, 其中一个激酶就是TFIIH的一个亚基。
RNA聚合酶II尾巴的磷酸化意义: 帮助RNA聚合酶摆脱起始时大部分通用转录因子, 也控制RNA的延伸和加工。
TBP用一个插入双螺旋小沟的β折叠来结合并扭曲DNA
TBP依赖TATA序列能够经受特殊的结构扭曲的能力而对其进行选择。A: T碱基对是更有利的, 因为它们更易于被扭曲而使小沟开始开放。
其他通用转录因子在起始中也有特殊作用
TAF: TBP与大约10个TAF相关。两个TAF在启动子处结合DNA元件, 如起始密码子元件(Inr)和下游启动子元件(DPE)。几个TAF与组蛋白有结构同源性。另一个TAF似乎调节TBP与DNA的结合。
TF I B: 此蛋白质为一条多肽单链, 在TBP之后进入前起始复合体。特殊的TF II B-TBP和TF II B-DNA联系间的碱基特异性相互作用。TF II B在前起始复合体中也与Pol II接触。因而, 此蛋白质似乎是连接结合了TATA的TBP与聚合酶之间的桥梁。TF II B的N端域与细菌中σ3.2相似的方式插入Pol II的RNA出口通道。TF II F: 这个双亚基因子与Pol II结合并与Pol II(以及其他因子)一起被募集到启动子上。Pol II与TF II F的结合起着稳定DNA-TBP-TF II B复合体的作用, 并且是TF II E和TF II H被募集到前起始复合体上的前提条件。
TF II E和TF II H: TF II E与TF II F相似, 由两个亚基构成, 它是下一个结合到前起始复合体上的, 并参与TF II H的募集以及调节。TF II H控制着依赖ATP的从前起始复合体向开放复合体的转变。TF II H内有两个亚基具有ATP酶的功能, 还有一个亚基是蛋白激酶, 参与启动子的解旋和逃离, 与其他因子一起, ATP酶亚基还参与核苷酸错误匹配的修复。
体内的转录起始需要其他蛋白质参与,包括中介蛋白复合体
由于DNA模板在细胞内包装到染色质内, 细胞内高水平的、受调节的转录还额外需要中介蛋白复合体和转录调节蛋白, 并在很多情况下需要核小体修饰酶(其自身常常是大的蛋白质复合体的组成部分)。
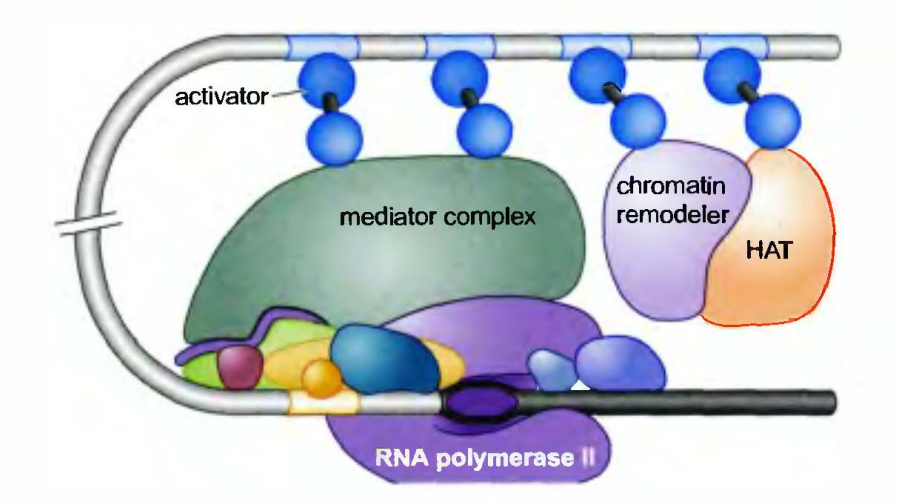
中介蛋白复合体: 含有多个亚基, 激活因字帮助聚合酶募集到启动子上, 使其稳定在那里, 募集过程的一个相互作用是与中介复合体进行, 因此而得名。其中一个表面与聚合酶的CTD尾巴结合, 其他表面与激活因字结合。
功能: 不同激活因字与中介蛋白的不同亚基结合将聚合酶II带到不同的基因上。调控TFIIH内的CTD激酶来帮助转录起始。
中介蛋白包含很多亚基,其中有些亚基从酵母到人类是保守的
酵母和人的中介蛋白各自包括20个以上的亚基, 其中7个在两种生物之间表现出显著的序列同源性。只有一个(Srb4)是几乎所有PolⅡ基因在细胞内转录所必需的。
酵母和人类的中介蛋白都是由一些模块组装而成的。一个由PolⅡ、中介蛋白和一些通用转录因子组成的复合体可以在DNA不存在的情况下座位一个单独的复合体从细胞中分离出来。预先形成的复合体被命名为RNA PolII全酶, 也因而能够起始转录。
一组新的因子激发PolII延伸和RNA校对
延伸阶段: 一旦聚合酶起始了转录, 便转入了延伸阶段。这一转变包括PolⅡ酶脱落其大部分起始因子, 如通用转录因子和中介蛋白。另一组因子被募集在它们的位置上。其中的一些(如TFⅡS和hSPT5)是延伸因子(elongation factor), 也就是激发延伸的因子, 其他的是RNA加工所需要的。
激酶P-TEFb, 它是由转录激活因子募集到聚合酶上的。一旦与PolⅡ结合, 此蛋白质就将CTD重复序列中位置2处的丝氨酸残基磷酸化。P-TEFb磷酸化并因此激活另一个称为hSPT5的蛋白质。还有一个延伸因子, TAT-SF1, 被P-TEFb募集。
另一个不影响起始但激发延伸的因子是TFⅡS。此因子促进延伸的总体速度, 这是通过在遭遇到趋向于减缓聚合酶进程的序列时限制其暂停时间而实现的。
TFⅡS还有另外一个功能: 它有助于由聚合酶进行的校对。
延伸RNA聚合酶在行进过程中必须应对组蛋白的空间阻碍
RNA聚合酶如何通过这些潜在的障碍而转录?
促进染色体转录(facilitates chromatin transcription, FACT)因子: 这个因子使染色质模板进行的转录更有效。FACT是两个非常保守的蛋白的异型双聚体(heterodimer)——Spt16和SSRP1。FACT在延伸中的作用是通过这个复合体与包括TFIIS在内的已知的延伸因子之间的遗传相互作用而建立的。
FACT如何起作用?
在转录中RNA聚合酶的前方, FACT挪开一个H2A.H2B二聚体。这使得聚合酶通过那个核小体(在体外, 已经显示了从模板上挪开一个H2A.H2B二聚体使转录能够进行)。FACT还有组蛋白伴护活性, 这使得它能够在行进的聚合酶后将H2A.H2B二聚体立即放回到组蛋白六聚体中。如此, FACT使得聚合酶延伸, 同时保持了转录中染色质的完整性。
延伸聚合酶与各种RNA加工所需要的一组新的蛋白质因子相互作用
这些加工事件包括下面几个: RNA的5’端加帽(capping)、剪接(splicing)和RNA的3’端的多聚腺苷酸化(polyadenylation)。转录的延伸、终止和RNA加工似乎是相互联系的。
第一个RNA加工事件是加帽, 即在RNA的5’端添加一个修饰过的鸟嘌呤碱基。加帽之后, 尾部序列重复中的Ser5发生去磷酸化而导致加帽机器的脱离, 而进一步的磷酸化(这次是尾部重复中的Ser2)又导致RNA剪接所需要的机器的募集。
一旦聚合酶到达基因的末端, 就会遇到一段特殊的序列, 此序列在转录到RNA之后会引发多聚腺苷酸化酶向该RNA的转移, 导致4个事件的发生:mRNA的切割、多个腺嘌呤被添加到其3’端、将与RNA聚合酶结合着的RNA按照由5’到3’核酸酶进行降解, 以及随后由聚合酶进行的转录终止。
最后一个RNA加工事件——mRNA 3’端的多聚腺苷酸化。聚合酶的CTD尾巴参与募集多聚腺苷酸化所必需的酶。有些序列一旦转录到RNA中, 就会引发这些因子向RNA的转移, 这些序列称为poly-A信号。一旦CPSF和CstF结合到RNA上, 其他蛋白也就被募集, 先引起RNA的切割, 然后是多聚腺苷酸化。多聚腺苷酸化是由poly-A聚合酶介导的, 此酶在由切割所形成的RNA的3’端添加大约200个腺嘌呤。(补充P478加尾机制)
- 5’端帽子功能:
- 保护mRNA不被核酸外切酶降解。
- 增加mRNA的可译性。
- 5, 端帽子帮助mRNA运输到细胞质, 作为翻译的模板。
- 通过5, 端帽子剪接第一个内含子。
- 使前体mRNA正确剪接。
- 3’多聚A尾巴功能:
- 通过3, 多聚A尾巴剪接最后一个内含子。
- 保护mRNA不被核酸外切酶降解。
- 增加mRNA的可译性。
转录终止模型
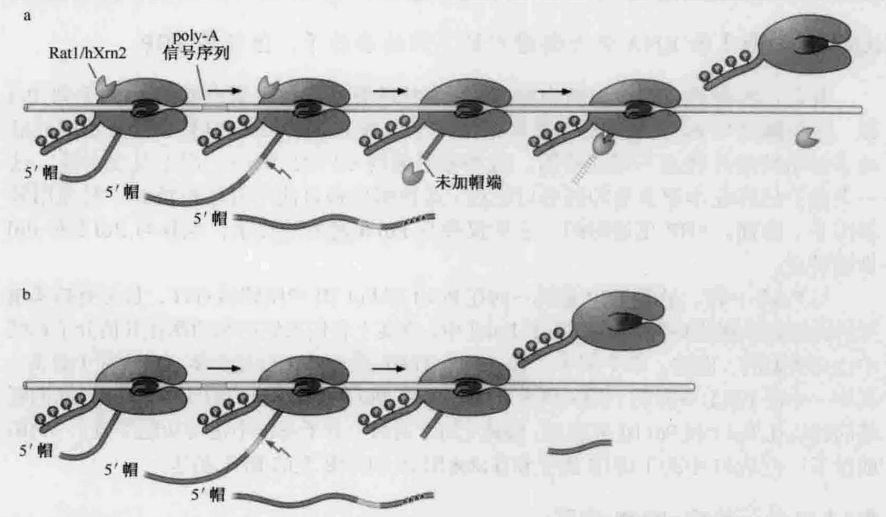
- a: 在鱼雷模型中(支持较多), poly-A位点下游转录的RNA被5’→3’RNase(鱼雷)攻击, 该RNase由聚合酶本身带到这个转录物上。当这个核酸外切酶追上聚合酶时, 它引发了从DNA模板上解离和转录的终止。
- b: 在变构模型中, 聚合酶在转录基因时具有高活性, 之后一旦过了poly-A位点, 活性降低。这个改变可能由于修饰和构象改变。即使在变构模型, 第二个RNA要被RNase降解, 但不是终止的原因。
由RNA聚合酶I和III催化的转录
RNA聚合酶Ⅰ和Ⅲ识别不同的启动子,使用不同组别的转录因子,但还是需要TBP
TBP是通用的, 它不仅参与PolII起始的转录, 也参与PolI和PolIII的转录。
PolI只用于转录rRNA基因
rRNA基因的启动子包括两个部分: 核心元件和UCE(上游控制元件, upstream controlelement)。除了PolI之外, 起始还需要另外两个称为SL1和UBF的因子。
PolIII启动子位于转录起始位点的下游
PolIII启动子种类:①tRNA基因的启动子②5SrRNA基因③snRNA基因PolIII进行的转录除需要聚合酶之外, 还需要转录因子。这些因子称为TFIIIB和TFIIIC(对于tRNA基因), 5S rRNA基因的转录还需要加上TFIIIA。